Metoda biochemiczna
Identyfikacja do rodzaju
Izolaty bakteryjne dające pozytywny wynik w testach na patogeniczność poddaje się najczęściej identyfikacji do rodzaju przy pomocy klucza biochemicznego opisanego np. takiego jak ten opisany w Schaad i wsp. (2001). Identyfikacja do rodzaju obejmuje wykonanie dla każdego izolatu następujących oznaczeń biochemicznych i fizjologicznych: barwienia Grama, testu na fermentację glukozy na pożywce Hugh - Leifson (HL), wytwarzania pigmentu na pożywce z węglanem wapnia (Yeast extract Dextrose Calcium agar (YDC)), wytwarzanie na pożywce King B pigmentu fluoryzującego w świetle UV i zdolność wzrostu na pożywce D1M.
Barwienie Grama wykonuje się w następujący sposób. Na utrwalony na szkiełku podstawowym preparat bakteryjny nanosi się równe ilości roztworu fioletu krystalicznego i kwaśnego węglanu wapnia, barwi przez 10 sek. i zlewa roztwór. Następnie na preparat nanosi się roztwór jodu, utrwala przez 10 sek., zlewa i dokładnie spłukuje wodą destylowaną. Do odbarwiania preparatu stosuje się roztwór acetonu i alkoholu etylowego (3:1), spłukuje i nanosi barwnik kontrastujący, fuksynę zasadową. Preparat ogląda się w mikroskopie świetlnym, przy powiększeniu 100x, pod olejkiem immersyjnym (Fig. 1.).
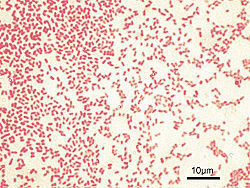
Fig. 1. Przykład barwienia Grama
Identyfikacja do gatunku
Testy API
Do wstępnej identyfikacji biochemicznej badanych izolatów bakteryjnych, wyizolowanych z roślin i spełniających postulaty Kocha, często używa się testów API (BioMerieux, Francja), a w szczególności testów API 20E i API 20NE. Testy API 20E stosowano do badania Gram-ujemnych, fermentujących pałeczek, natomiast testy API 20NE zostały użyte do charakterystyki Gram-ujemnych, niefermentujących pałeczek.
Test API 20E (bioMerieux, Francja) jest gotowym zestawem diagnostycznym zawierającym sproszkowane pożywki do identyfikacji fermentujących pałeczek Gram-ujemnych, umożliwiającym oznaczenie w jednym badaniu 21 cech biochemicznych badanego izolatu. Test ten obejmuje następujące oznaczenia biochemiczne: aktywność enzymów: ß-galaktozydazy, dihydrolazy argininy, dekarboksylazy lizyny, dekarboksylazy ornityny, ureazy, dezaminazy tryptofanu, żelatynazy, oksydazy cytochromowej, wykorzystanie cytrynianu, wytwarzanie H2S, indolu i acetoiny (Voges Proskauer), fermentacja wielocukrów: glukoza, mannitol, inozytol, sorbitol, ramnoza, sacharoza, melibiozy, amigdalina, arabinoza.
Test API 20NE (BioMerieux, Francja) jest gotowym zestawem diagnostycznym zawierającym sproszkowane pożywki do identyfikacji niefermentujących pałeczek Gram-ujemnych, umożliwiającym oznaczenie w jednym badaniu 21 cech biochemicznych badanego izolatu. Test ten obejmuje następujące oznaczenia biochemiczne: redukcja azotanów do azotynów, redukcja azotynów do azotu, wytwarzanie indolu, fermentacja glukozy, aktywność enzymów: dihydrolazy argininy, ureazy, ß-glukozydazy, proteazy, ß-galaktozydazy, oksydaza cytochromowa, asymilacja węglowodanów: glukoza, arabinoza, mannoza, mannitol, N-acetylo-glukozamina, maltoza, glukonian potasu, kwas dekanowy, kwas adypinowy, jabłczan, cytrynian trisodowy, kwas fenylooctowy.
Wyniki, uzyskane w postaci siedmiocyfrowych kodów API, reprezentujących profile biochemiczne badanych izolatów, porównywano z bazą kodów dostarczoną przez producenta testów oraz z analogicznymi kodami szczepów referencyjnych bakteryjnych patogenów roślin opisanych w literaturze (Mergaert i wsp. 1984, Mergaert i wsp. 1993, Lamka i wsp. 1991, Vantomme i wsp. 1986). Dodatkowo, jako szczepy porównawcze do testów API wykorzystano bakteryjne izolaty referencyjne.
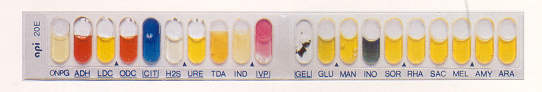
Fig.2. Przykład testu API 20E.
System BIOLOG
Jako potwierdzenie i rozszerzenie wstępnej identyfikacji biochemicznej uzyskanej przy pomocy testów API, stosuje się system BIOLOG (BIOLOG Inc., Hayward, CA). Jest to gotowy zestaw zawierający sproszkowane podłoża służące do identyfikacji bakterii Gram-dodatnich i Gram-ujemnych. System ten umożliwia jednoczesne oznaczenie 95 cech biochemicznych badanego izolatu bakteryjnego tworząc jego unikalny profil biochemiczny, który po odczytaniu i wprowadzeniu do bazy programu jest porównywany przy użyciu specjalnie opracowanego algorytmu z blisko 3 tysiącami innych profili biochemicznych, w tym z profilami bakterii fitopatogenicznych. Algorytm ten, na podstawie różnic między poszczególnymi profilami biochemicznymi oszacowuje stopień pokrewieństwa badanych izolatów i wyraża go w specjalnych jednostkach. Na tej podstawie następuje identyfikacja badanej bakterii i podawane są również nazwy gatunków o najbardziej zbliżonym profilu biochemicznym.
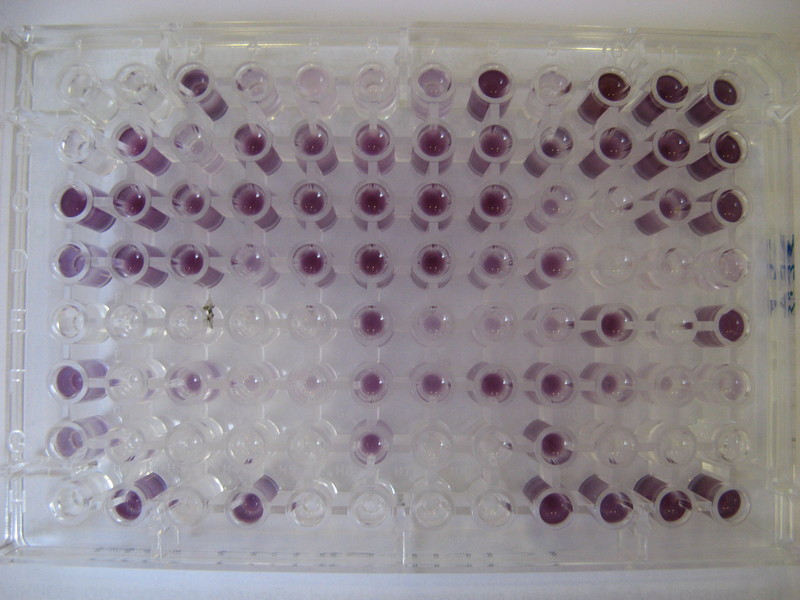
Fig. 3. Przyład testu Biolog GEN III.
Analiza kwasów tłuszczowych (FAME)
Inną często stosowaną biochemiczną metodą identyfikacji bakterii jest analiza profilu estrów metylowych kwasów tłuszczowych (ang. fatty acid methyl analysis – FAME). Metoda ta jest stosowana od ponad 50 lat. Dotychczas w trakcie badań odkryto istnienie w błonie cytoplazmatycznej i w otoczce zewnętrznej (u bakterii Gram ujemnych), ponad 300 różnych kwasów tłuszczowych i ich pochodnych. Odkryto również, że cechą diagnostyczną wielu gatunków bakterii jest nie tylko skład kwasów tłuszczowych, ale również ich ilość. Dlatego też kombinacja ponad 300 różnych kwasów tłuszczowych z ich ilościowym występowaniem daje duże możliwości w diagnostyce bakterii, ponieważ niemal każdy gatunek z posiada swój specyficzny profil kwasów tłuszczowych. Analiza kwasów tłuszczowych polega na ich ekstrakcji z badanych mikroorganizmów, przekształceniu do form estrowych i następnie identyfikacji ich lotnych pochodnych w chromatografie gazowym. Otrzymuje się w ten sposób pewien wzór pików, który jest charakterystyczny dla danego gatunku mikroorganizmu i możne być porównany z innymi wzorami zgromadzonymi w bazie danych programu. Metoda ta jest szeroko stosowana do charakterystyki nowo odkrytych patogenów oraz do identyfikacji szczepów patogenicznych.

Fig. 4. Przykład wyników analizy kwasów tłuszczowych (FAME)
Literatura:
Mergaert J., Verdonck L., Kersters K., Swings J., Boeufgras J. M., De Ley J. 1984. Numerical taxonomy of Erwinia species using API sytems. Journal of General Microbiology 130: 1893-1910.
Mergaert J., Verdonck L., Kresters K. 1993. Transfer of Erwinia ananas (synonym, Erwinia uredovora) and Erwinia stewartii to the genus Pantoea emend. as Pantoea ananas (Serrano 1928) comb. nov. and Pantoea stewartii (Smith 1898) comb. nov., respectively, and description of Pantoea stewartii subsp. indologenes subsp. nov. International Journal of Systematic Bacteriology, 43: 162-173.
Lamka G. L., Hill J. H., McGee D. C., Braun E. J. 1991 Development of an immunosorbent assay for seedborne Erwinia stewartii in corn seeds. Phytopathology, 81: 839-846.
Vantomme R., Rickaert C., Swings J., De Ley J. 1986. Characterization of further Erwinia amylovora strains and the application of the API 20E system in diagnosis. J. Phytopath., 117: 34-42.