Techniki molekularne stosowane do wykrywania agrofagów
Najpopularniejszą techniką biologii molekularnej wykorzystywaną w naszym laboratorium jest reakcja łańcuchowa polimerazy (ang. polymerase chain reaction, PCR). Większość wirusów roślin, z którymi pracujemy, posiada materiał genetyczny w postaci RNA stąd też często jest ona poprzedzona reakcją odwrotnej transkrypcji, umożliwiającą uzyskanie cDNA z wyizolowanego RNA. Reakcja PCR składa się z wielokrotnie powtarzanego cyklu trzech etapów, które zachodzą w różnych temperaturach: denaturacji, przyłączania starterów i syntezy nici DNA. Reakcja przebiega w termocyklerze. Po reakcji konieczny jest rozdział elektroforetyczny uzyskanych produktów w żelu agarozowym względem markera wielkości mas. W reakcji PCR niezwykle istotny jest dobór odpowiednich starterów, które będą umożliwiały wykrycie jak najszerszego zakresu izolatów wirusa często zróżnicowanych genetycznie. Ważna jest również specyficzność reakcji: startery powinny być tak zaprojektowane, aby nastąpiła wyłącznie amplifikacja konkretnego genu lub rejonu w genomie agrofaga.
Jednym z rodzajów reakcji PCR jest PCR w czasie rzeczywistym (qPCR, ang. quantitative PCR). Technika ta, dzięki zastosowaniu barwników fluorescencyjnych lub sond pozwala na monitorowanie ilości produktu reakcji trakcie jej trwania i tym samym pozwala wyeliminować etap elektroforezy po zakończeniu reakcji. Stosowanie sond typu TaqMan, będących krótkimi oligonukleotydami zawierającymi na końcu 5’ znacznik fluorescencyjny, a na końcu 3’ jej wygaszacz, zwiększa czułość i specyficzność reakcji. Niektóre termocyklery do reakcji qPCR dają również możliwość przeprowadzanie analizy profilu topnienia (denaturacji) DNA po reakcji (ang. high resolution melt). Technika ta umożliwia wykrywanie mutacji, polimorfizmów, małych delecji lub insercji. Pozwala to na szybkie wykrycie np. patotypów wirusa, które różnią się pojedynczą mutacją w danym regionie genomu.
W ostatnim czasie niezwykłą popularnością cieszy się izotermalna amplifikacja kwasów nukleinowych metodą przemieszczających się pętli w stałej temperaturze (z ang. Loop-mediated isothermal AMPlification, LAMP) http://www.novazym.pl/images/32-37.pdf. Technika ta polega na specyficznej syntezie nowych nici DNA na matrycy w kolejnych cyklach reakcji oraz na zastępowaniu łańcuchów DNA przez nici nowo zsyntetyzowane. Reakcja przebiega w stałej temperaturze (zwykle 60-65°C). Ponadto, dodanie odpowiednich barwników fluorescencyjnych do reakcji powoduje zmianę jej zabarwienia z pomarańczowej na zieloną w świetle UV w przypadku wyniku pozytywnego i wykrycia patogena, w związku z czym metoda nie wymaga wizualizacji produktów reakcji w żelu agarozowym. Technika LAMP pozwala nie tylko na bardzo szybkie (nawet w 15 minut) wykrycie patogena, ale jest również bardzo czuła. Zastosowanie 3 par starterów zwiększa jej specyficzność. Ze względu na fakt, że nie wymaga ona stosowania specjalistycznego sprzętu może być wykorzystywana do wykrywania patogenów bezpośrednio w warunkach polowych lub szklarniowych.
W Klinice Chorób Roślin opracowaliśmy szereg różnych metod pozwalających na szybkie i skuteczne wykrywanie różnych gatunków wirusów, a także rozróżnianie ich wariantów genetycznych.
WYBRANE PUBLIKACJE (2013-2015)
1. Hasiów-Jaroszewska B., Borodynko N. 2013. Detection of Pepino mosaic virus isolates from tomato by one-step reverse transcription loop-mediated isothermal amplification. Archives of Virology 158 (10): 2153-2156.
2. Hasiów-Jaroszewska B., Komorowska B. 2013. A new method for detection and discrimination of Pepino mosaic virus isolates using high resolution melting analysis of the triple gene block 3. Journal of Virological Methods 193: 1-5.
3. Hasiów-Jaroszewska B, Rymelska N. Borodynko N, 2015. LNA probe-based assay for the detection of Tomato black ring virus isolates. Molecular and Cellular Probes 29(1):78-80.
Bio-PCR
Bio-PCR jest odmianą techniki PCR w której czułość standardowej reakcji PCR wzbogacona została o etap wstępnej hodowli bakterii. Polega to na wysianiu soku roślinnego z potencjalnie zakażonej rośliny, na podłoże mikrobiologiczne i jego 24 godzinnej hodowli celem podniesienia liczebności komórek bakteryjnych. Po zakończeniu inkubacji wszystkie wyrosłe na podłożu mikroorganizmy są z niego zmywane i poprzez gotowanie przygotowywana jest z nich matryca DNA do reakcji PCR. Dzięki temu etapowi komórki patogena występujące w roślinie nielicznie i nie wywołujące jeszcze symptomów chorobowych, mogą zostać namnożone, a przez to ich obecność zostanie wykazana w reakcji PCR. Dlatego Bio-PCR jest doskonałym uzupełnieniem konwencjonalnej reakcji PCR.
PCR ze specyficznymi starterami
Na potrzeby diagnostyki molekularnej bakterii opracowano i przetestowano startery do reakcji PCR, zaprojektowane na podstawie rejonów genomu charakterystycznych dla badanego przez nas gatunku. Efektem takiej reakcji najczęściej jest otrzymanie właściwej wielkości produktu dla pożądanego przez nas gatunku i jego brak dla innych gatunków. Przykładem tego typu doświadczenia jest wykrywanie za pomocą starterów specyficznych bakterii Pectobacterium carotovorum subsp. carotovorum (Darrase i wsp. 1994), Pantoea stewartii subsp. stewartii (De Boer 2001, Coplin i wsp. 2002, Carlier i von Bodman 2006), P. ananatis (Gitaitis i wsp. 2009, Walcott i wsp. 2002, De Boer 2001) oraz P. agglomerans (Schaad i wsp. 2001).
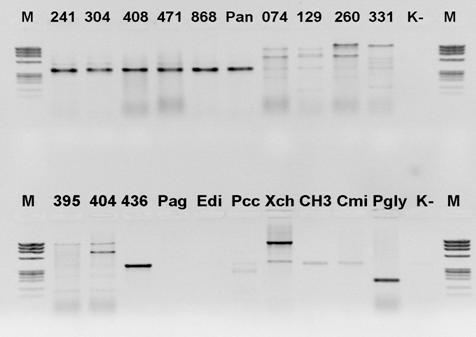
Fig.1. Przykład specyficznego działania starterów do reakcji PCR.
Genotypowanie
Genotypowaniem określa się zastosowanie technik molekularnych wykorzystujących m.in. startery projektowane na potrzeby danego badania to techniki wykorzystujące wielokrotne, krótkie, wewnątrzgenomowe powtórzenia sekwencji. Zalicza się tu techniki: ERIC, REP i BOX. Sekwencje ERIC (ang. Enterobacterial Repetitive Intergenic Consensus) są niedoskonałymi sekwencjami palindromicznymi, o długości 127 pz, występującymi w licznych kopiach w genomie bakterii z rodziny Enterobacteriaceae. Rozmieszczenie tych powtórzeń w obrębie genomu bakterii jest jej cechą charakterystyczną, na której opiera się istota identyfikacji tą techniką. Badanie polega na przeprowadzeniu reakcji PCR ze starterami komplementarnymi do sekwencji ERIC i następnie na rozdziale produktów PCR na żelu agarozowym. Ilość i układ uzyskanych w ten sposób prążków jest cechą charakterystyczną dla poszczególnych gatunków lub nawet szczepów. Porównanie ortologicznych kopii pomiędzy gatunkami Enterobacteriaceae wykazało, że sekwencje ERIC są zaskakująco konserwatywne, co wskazuje na to, że musiały one pełnić wcześniej określone funkcje w komórce, najprawdopodobniej związane ze stabilnością mRNA. Na tej samej zasadzie co sekwencje ERIC do genotypowania wykorzystywane są również inne typy powtórzeń sekwencji w genomach bakterii, takie jak: BOX – wysoce konserwowane, krótkie, powtórzone regiony zlokalizowane, w obrębie międzygenowego DNA i REP (ang. Repetitive Extragenic Palindromic sequences).
Inna, enzymomolekularna grupa technik molekularnych wykorzystuje enzymy restrykcyjne. W grupie tej wyróżnić można trzy podstawowe techniki: RAPD (ang. Randomly Amplified Polimorphic DNA), RFLP (ang. Restriction Fragments Length Polymorphism) i AFLP (ang. Amplified Fragments Length Polymorphism). W metodzie RAPD wykorzystuje się kilka (zwykle 2-4) starterów, które w reakcji PCR łączą się w różnych miejscach do genomu badanej bakterii. Po zakończeniu reakcji powstaje mieszanina różnej wielkości produktów PCR które rozdziela się i uwidacznia w czasie rozdziału elektroforetycznego. Technika RFLP polega na trawieniu wybranego fragmentu (lub całego) genomu bakteryjnego jednym lub więcej enzymami restrykcyjnymi i elektroforetycznym rozdziale uzyskanych fragmentów DNA. Istotą obu wyżej wymienionych technik jest uzyskanie profilu prążków DNA rozdzielonych elektroforetycznie, które to profile można porównywać między badanymi gatunkami czy izolatami bakterii. Tym, co powoduje różnice w uzyskanych profilach elektroforetycznych jest występowanie u poszczególnych gatunków mutacji punktowych, które powodują nieprzyłączenie się startera w danym miejscu genomu (RAPD), bądź też zniknięcie lub pojawienie się nowego miejsca restrykcyjnego (RFLP).
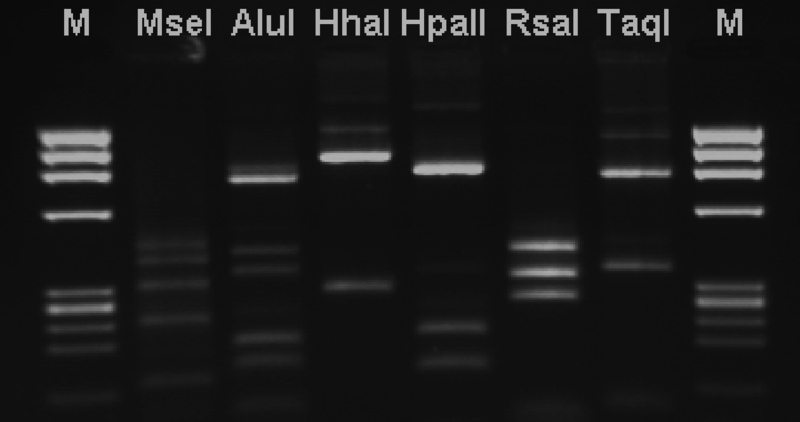
Fig.2. Przykład wyników uzyskanych techniką RFLP.
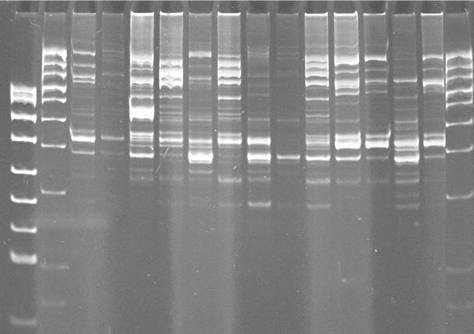
Fig. 3. Przykład wyników genotypowania szczepów bakterii starterami BOX.
Sekwencjonowanie 16S rRNA
Najszerzej stosowaną i należącą dziś do standardowych technik molekularnych jest sekwencjonowanie i analiza sekwencji nukleotydowych genów chodzących w skład rybosomalnego operonu 16S – ITS – 23S rRNA. Operon ten składa się z genu 16S rRNA, którego sekwencja jest wysoce konserwatywna w obrębie rodziny Enterobacteriaceae. Kolejnymi elementami tego operonu jest gen 23S rRNA, również mocno konserwowany w obrębie tej rodziny oraz znajdujący się pomiędzy tymi genami odcinek 16S – 23S ITS (ang. Internal Transcribed Spacer). Jest to rejon genomu, który podlega transkrypcji, ale już nie translacji. Dlatego też nie działa na niego presja selekcyjna i mutacje losowe, powstające w całym genomie mogą w tym akurat odcinku się kumulować gdyż nie wpływają one na funkcjonowanie żadnych produktów białkowych w komórce bakteryjnej. Dlatego też rejon ten jest wysoce zmienny i nadaje się do identyfikacji bakterii na poziomie gatunków i nawet podgatunków. Na podstawie tego odcinka stworzona została pierwotnie klasyfikacja P. ananatis, jednak, kiedy zaczęto amplifikować odcinek 16S – 23S rRNA używając uniwersalnych starterów, okazało się, że bakterie posiadają w swoim genomie po kilka kopii tego operonu. Oznacza to, że rejon ten może być nadal podstawą identyfikacji bakterii, natomiast nie można już na nim opierać analiz filogenetycznych.
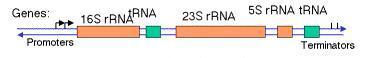
Fig.4. Schemat operonu bakteryjnego 16S rRNA.
MLSA
Techniką, która może zastąpić sekwencjonowanie 16S rRNA, jako podstawę identyfikacji i analiz filogenetycznych jest MLSA (ang. Multi Locus Sequence Analysis). Została ona opracowana do szybkiej identyfikacji i klasyfikacji bakterii z rodzaju Pantoea, i polega ona na uzyskaniu sekwencji kilku tzw. genów metabolizmu podstawowego (ang. house-keeping genes) (np. gyrB, rpoB, atpD, infB) i jednoczesnej ich analizie przy użyciu odpowiednich programów filogenetycznych. Technikę tę wykorzystano również do analizy zróżnicowania genetycznego w obrębie kompleksu Enterobacter cloaceae.
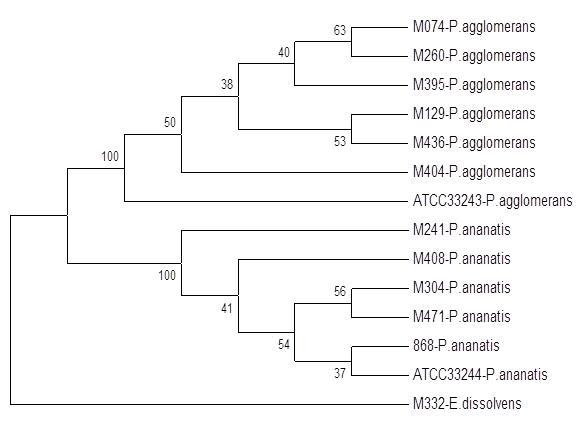
Fig.5. Przykład wyników uzyskanych techniką MLSA.