Wirus mozaiki ogórka na cukinii
Wirus mozaiki ogórka (Cucumber mosaic virus) należy do rodzaju Cucumovirus w rodzinie Bromoviridae. Wszystkie wirusy należące do tego rodzaju posiadają genom w postaci trzech jednoniciowych cząsteczek RNA o dodatniej polarności. Nici RNA 1 (długość ≈ 3357 nt), RNA 2 (długość ≈ 2947 nt) i RNA 3 (długość ≈ 2188 nt) o odpowiednio malejących masach cząsteczkowych, są pakowane pojedynczo w izometryczne cząstki o średnicy około 28 nm, o ikozaedralnej symetrii. CMV poraża około 1200 gatunków roślin, a wśród jego naturalnych gospodarzy znajdują się rośliny dyniowate – cukinia, ogórek, dynia. Wirus przenoszony jest przez różne gatunku mszyc. W Polsce zaobserwowano nasilone występowanie wirusa w uprawach cukinii, na której wirusy powodował różnego typu objawy: mozaiki, chlorozy , deformacje liści a nawet nekrozy (Fig. 1 A, B). Do wykrywania wirusa wykorzystywaliśmy technikę RT-PCR oraz startery amplifikujące region białka płaszcza (Tabela 1).


Fig. 1 A) Mozaiki na cukinii porażonej CMV B) Deformacja liści i nekrozy powodowane przez CMV
Tabela 1. Startery wykorzystywane do wykrywania CMV.
|
Starter |
Sekwencja 5’-3’ |
Temp. topnienia |
|
CMV CPf |
GCCGTAAGCTGGATGGAC |
53℃ |
|
CMV CPr |
GCTTCTCCGCGAG |
53℃ |
Cechą charakterystyczną wirusa jest wysoki stopień zróżnicowania biologicznego i genetycznego. Izolaty CMV podzielono na grupy IA, IB, II oraz III na podstawie wywoływanych przez nie objawów oraz podobieństwa sekwencji. W Polsce stwierdziliśmy występowanie izolatów z grupy IA oraz II (Fig. 2). Analizy filogenetyczne przeprowadziliśmy na podstawie genu kodującego białko płaszcza (CP).
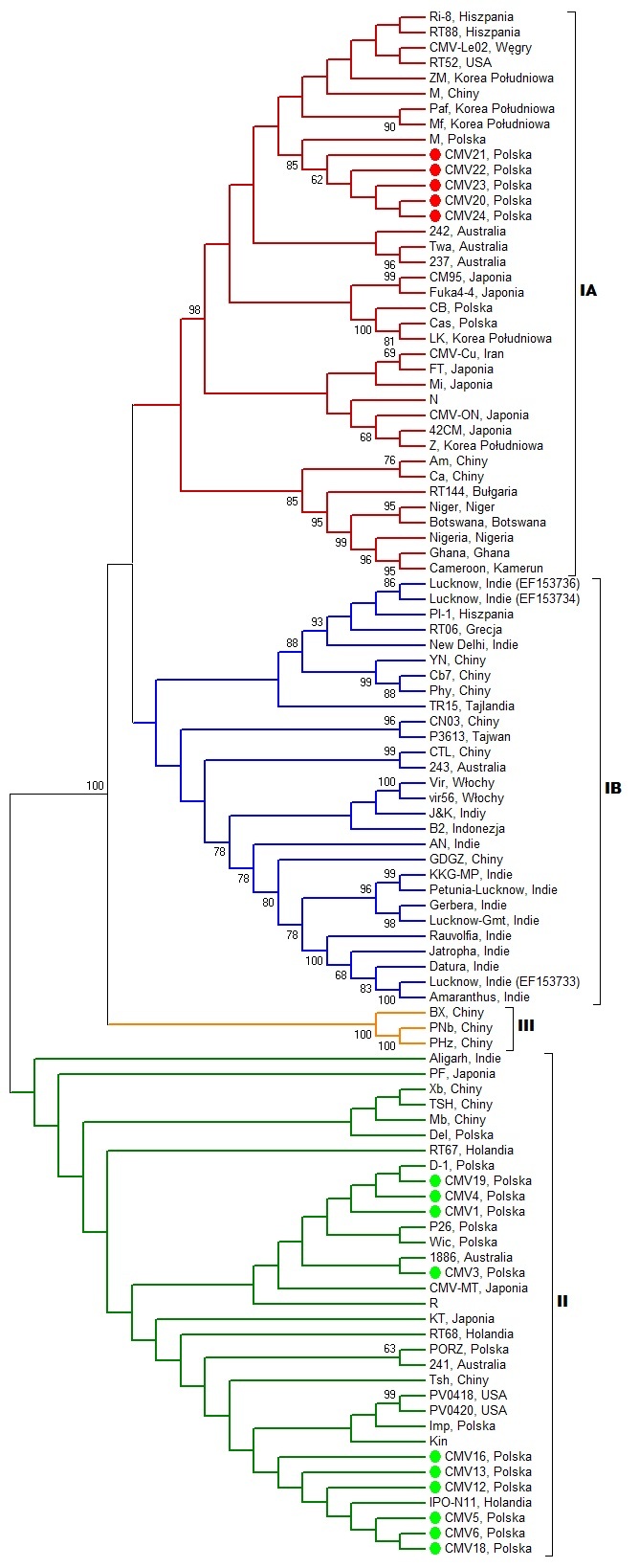
Fig. 2. Drzewo filogenetyczne skonstruowane w programie MEGA6 z wykorzystaniem sekwencji genu kodującego białko płaszcza polskich izolatów CMV i innych, których sekwencje zdeponowano w Banku Genów. Polskie izolaty oznaczono na czerwono i zielono.
Wysoki stopień zróżnicowania genetycznego CMV w istotny sposób wpływa na efektywność jego wykrywania, dlatego tak ważne jest dobranie odpowiedniej metody diagnostycznej w celu kontroli materiału roślinnego. W Klinice Chorób Roślin opracowaliśmy metodykę wykrywania genetycznie różnych izolatów CMV za pomocą izotermalnej amplifikacji kwasów nukleinowych (LAMP). Technika ta umożliwia wykrycie izolatów należących do dwóch różnych grup filogenetycznych w warunkach stałej temperatury i czasie krótszym niż godzina zegarowa (Fig. 3 A, B). Obecnie trwają prace nad możliwością stosowania tej metody w warunkach polowych.
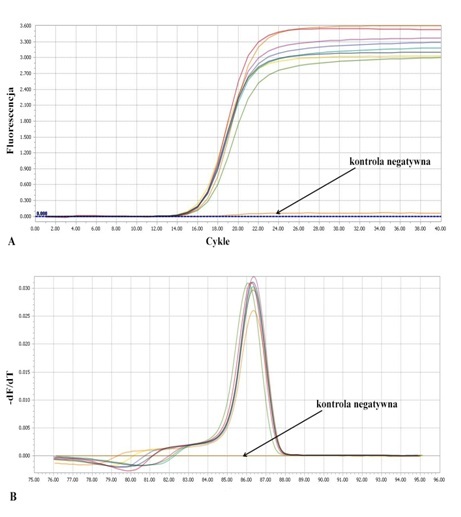
Fig. 3 A) Krzywe amplifikacji i B) dysocjacji otrzymane w wyniku reakcji LAMP. Do reakcji wykorzystano izolaty CMV z I i II grupy.