Wirus mozaiki pepino - zmienność i diagnostyka.
Wirus mozaiki pepino (Pepino mosaic virus, PepMV) jest obecnie jednym z najgroźniejszych patogenów pomidora szklarniowego w wielu krajach. Jest on typowym reprezentantem wirusów z rodzaju Potexvirus, zarówno jeśli chodzi o organizację genomu (Fig. 1), jak i morfologię cząstek wirusa (Fig. 2). Ze względu na swoją szkodliwość, łatwość rozprzestrzenia się i straty, jakie powoduje w ilości, i jakości plonu został on umieszczony na liście alertowej EPPO (European and Mediterranean Plant Protection Organization – Europejska i Śródziemnomorska Organizacja Ochrony Roślin). Opisane dotąd izolaty PepMV różnią się genetycznie, co było podstawą wyodrębnienia czterech genotypów wirusa: peruwiańskiego (LP), europejskiego (EU), amerykańskiego 1 (US1/CH1) oraz chilijskiego 2 (CH2).

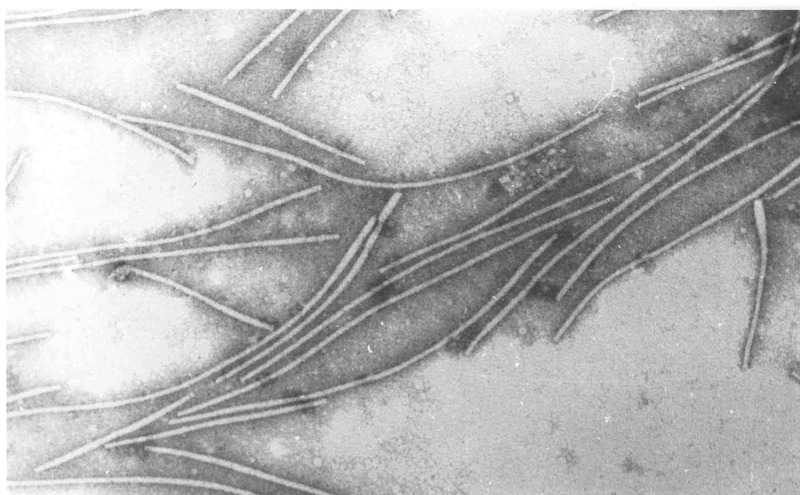
Fig. 1. Schemat organizacji genomu PepMV Fig. 2. Cząstki wirusa w transmisyjnym mikroskopie elektronowym
Zróżnicowanie biologiczne wirusa
W Polsce również dominują izolaty z grupy CH2, aczkolwiek odnotowywano również obecność innych genotypów. Prowadzone na szeroką skalę badania nad wirusem mozaiki pepino pozwoliły wykazać istotne różnice między izolatami zarówno genetyczne, jak i dotyczące objawów powodowanych na roślinach i zasięgu roślin żywicielskich. W obrębie genotypu CH2 stwierdziliśmy występowanie trzech patotypów wirusa, powodujących różnego typu objawy na infekowanych roślinach pomidora: łagodny, często niepowodujący objawów na roślinach, nekrotyczny – wywołujący nekrozy na roślinach oraz żółtaczkowy, który powoduje żółte zabarwienie blaszki liściowej (Fig. 3). Wirusa często izolowano z roślin ze słabymi objawami mozaiki lub brakiem wyraźnych objawów na liściach oraz z owoców z objawami marmurkowatości spowodowanej nierównomiernym wybarwieniem (Fig. 4).


Fig. 3. Roślina pomidora porażona PepMV A) izolat łagodny B) nekrotyczny C) żółtaczkowy Fig. 4. Owoce pomidora porażone PepMV
Zróżnicowanie genetyczne wirusa
Populacja wirusa jest bardzo zróżnicowana a izolaty reprezentujące różne genotypy wykazują od 75-100% podobieństwa sekwencji. Prowadzone do tej pory badania pozwoliły zakwalifikować polskie izolaty do grupy EU i CH2. Obecnie powszechnie dominują izolaty z grupy CH2, które wykazują wysokie podobieństwo sekwencji, ale cechują się zupełnie odmiennymi właściwościami biologicznymi. Wirus tworzy również populacje typu pseudogatunku. W toku badań uzyskano pełne sekwencje różnych patotypów wirusa, które są zdeponowane w Banku Genów http://www.ncbi.nlm.nih.gov pod numerami: nekrotyczny - HQ650559.1 http://www.ncbi.nlm.nih.gov/nuccore/328879871, łagodny - HQ650560.2 http://www.ncbi.nlm.nih.gov/nuccore/449042834 i żółtaczkowy - JX417070.1 http://www.ncbi.nlm.nih.gov/nuccore/406857467.
Genetyczne determinanty zmienności
Uzyskanie w 2009 infekcyjnych kopii wirusa i wykorzystanie techniki ukierunkowanej mutagenezy umożliwiło rozpoczęcie zakrojonych na szeroką skalę badań nad identyfikacją i analizą genetyczną nowopowstających w naturze patotypów wirusa. Badania wykazały, że pojedyncza substytucja w kodonie A199AA izolatów łagodnych na G199AA u nekrotycznych powodowała zmianę zasadowej lizyny (K) w kwas glutaminowy (E) w pozycji 67 TGB3 i wpływa na rozwój nekrotycznych symptomów na roślinach pomidora (Hasiów-Jaroszewska i wsp. 2011, Hasiów-Jaroszewska, Borodynko 2012).
Badania wykazały, że dwie zmiany reszt aminokwasów (E155K oraz D166G) w białku płaszcza wpływają na rozwój żółtaczkowych objawów na roślinach pomidora wszystkich testowanych odmian. Analiza struktury trzeciorzędowej białka płaszcza wykonana z wykorzystaniem algorytmu Rosetta wykazała, że zarówno aminokwas 155, jak i 166 są zlokalizowane na powierzchni białka i tym samym mogą odgrywać rolę w oddziaływaniach białko-białko (Hasiów-Jaroszewska i wsp. 2013).
Diagnostyka
Do wykrywania PepMV opracowano szereg metod opartych na technice PCR: RT-PCR, IC-RT-PCR, real-time RT-PCR (Fig. 5). W reakcji real-time PCR najczęściej stosowane są startery opracowane przez Ling i wsp. (2007) Tabela 1.
Tabela 1. Startery i sonda użyte do detekcji PepMV w reakcji real-time RT-PCR
|
Starter |
Sekwencja 5’-3’ |
Amplifikowany typ izolatu |
|
KLO5-48 KLO5-51 KLO5-49 KLO5-52 KLO5-50 (SONDA) |
ACTCCTAGAGCTGACCTCAC TCTCCAGCAACAGGTTGGTA ACTCCTAGAGCTGATCTTAC TCACCTGCAACTGGTTGATA FAM-TGTCAGCTTGCATTTACTTCCAAAA-TAMRA |
Ch1, US1, EU, LP Ch1, US1, EU, LP Ch2, US2 Ch2, US2 Wszystkie |
W Klinice Chorób Roślin stosujemy technikę RT-PCR ze starterami amplifikującymi region TGB: TGB3 F: 5’GGTGGACAATATCAAGACCGG3’ i TGB3 R: 5’CTGTATTGGGTTTGAGAAGTC3’. W 2013 roku opracowaliśmy technikę izotermalnej amplifikacji kwasów nukleinowych (LAMP) do wykrywanie genetycznie różnych izolatów wirusa (Hasiów-Jaroszewska, Borodynko 2013). Pozwala ona na skuteczne wykrywanie izolatów reprezentujących różne genotypy w warunkach stałej temperatury w czasie 30 minut. Zastosowanie odpowiednich barwników, które dodawane są do reakcji umożliwia wizualizację w świetle UV bez potrzeby rozdziału produktów w żelu agarozowym. W przypadku pozytywnego wyniku i wykrycia wirusa następuje zmiana zabarwienia z pomarańczowej na zieloną (Fig. 6).
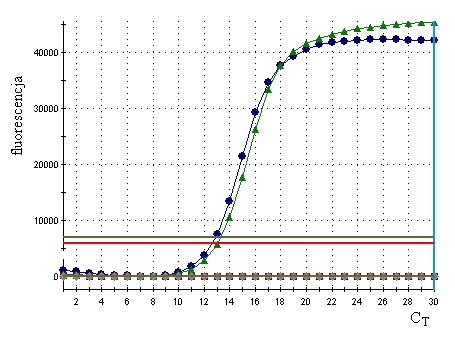

Fig. 5. Wykrywanie PepMV za pomocą real-time PCR Fig. 6. Wykrywanie PepMV za pomocą RT-LAMP.
Literatura
1. Hasiów-Jaroszewska B., Borodynko N., Jackowiak P., Figlerowicz M., Pospieszny H. 2011. Single mutation converts mild pathotype of the Pepino mosaic virus into necrotic one. Virus Research 159: 57-61.
2. Hasiów-Jaroszewska B., Borodynko N. 2012. Characterization of the necrosis determinants of the European genotype of pepino mosaic virus by site specific mutagenesis of an infectious cDNA clone. Archives of Virology 157: 337–341.
3. Hasiów-Jaroszewska B., Borodynko N. 2013. Detection of Pepino mosaic virus isolates from tomato by one-step reverse transcription loop-mediated isothermal amplification. Archives of Virology 158 (10): 2153-2156.
4 . Hasiów-Jaroszewska B., Paeleman A., Ortega-Parra N., Borodynko N., Minicka J., Czerwoniec A., Thomma BPHJ, Hanssen IM. 2013. Ratio of mutated versus wild-type coat protein sequences in Pepino mosaic virus determines the nature and severity of yellowing symptoms on tomato plants. Molecular Plant Pathology 14(9):923-933
5. Ling K.S., Wechter W.P., Jordan R. 2007. Development of a one-step immunocapture real-time TaqMan RT-PCR assay for the broad spectrum detection of Pepino mosaic virus. Journal of Virological Methods 144, 65-72.