Wirus Y ziemniaka na pomidorze
Wirus Y ziemniaka
Zróżnicowanie biologiczne
(Potato virus Y, PVY) należący do rodziny Potyviridae jest groźnym patogenem, szczególnie dla upraw o dużym znaczeniu gospodarczym, jak ziemniak, tytoń, pomidor czy papryka. Wirus jest przenoszony przez mszyce. Powoduje zróżnicowane objawy chorobowe, zależne od izolatu czy rośliny gospodarza, od łagodnych symptomów poprzez silne mozaiki, pofałdowania blaszek, nierównomierne wybarwianie się owoców aż po systemiczne nekrozy blaszek liściowych i owoców, opadanie liści oraz zahamowanie wzrostu. Istnieją trzy główne szczepy tego wirusa (PVY0, PVYN, PVYC) oraz liczne rekombinanty między nimi: PVYNTN, PVYZ, PVYE, PVYNWi, PVYNN242 oraz PVYNWi-P. W latach 2012-2014 na pomidorze gruntowym i szklarniowym stwierdzono liczne ogniska jego występowania. Co więcej zaobserwowano nie tylko obecność izolatów PVYNWi, ale również izolatów należących do szczepów PVY0 i PVYNTN. Obserwowano różnego typu objawy: zahamowanie wzrostu roślin, przewężenia liści, podwijanie blaszek liściowych (Fig. 1). W szklarniach natomiast na roślinach występowały mozaiki (Fig. 2).


Fig. 1 Roślina porażona PVY (pole) Fig. 2 Roślina porażona PVY (szklarnia)
Diagnostyka
Do wykrywania wirusa często stosowana jest technika RT-PCR ze starterami opracowanymi przez Rigotti i Gugerli (2007) (Tabela 1) pozwalającymi na zakwalifikowanie izolatów do odpowiednich szczepów. W Klinice Chorób Roślin opracowaliśmy również technikę izotermalnej amplifikacji kwasów nukleinowych (ang. loop-mediated isothermal amplification, LAMP), która umożliwia wykrycie wirusa w warunkach stałej temperatury w czasie krótszym niż godzina zegarowa. W technice tej wykorzystuje się 3 pary starerów co zwiększa jej specyficzność (Tabela 1) (Hasiów-Jaroszewska i wsp. 2015).
| Starter | Sekwencja 5’-3’ | Temperatura topnienia |
| PVY3+ | TGTAACGAAAGGGACTAGTGCAAAG | 57ºC |
| PVY3- | CCGCTATGAGTAAGTCCTGCACA | |
| PVYc3 | CAACGCAAAAACACTCA(CT)AA(AC)GC | |
| PVYf | TAAGTG(AG)ACAGACCCTCT(CT)TTCTC | |
| PVYCP2+ | CCAGTCAAACCCGAACAAAGG | |
| PVYCP1- | GGCATAGCGTGCTAAACCCA | |
| FIP | GCGCATTTCTATATACGCTTCACCAACC CTTAGGCAAATCAT | 63ºC |
| BIP | CGCAGCATTGAAATCAGCTGTGTTCTCC TCTTGTGTACTG | |
| LoopF | TGCAACATCTGAGAAATGTGC | |
| LoopB | CTCGACTTTTCGGGTTGGA | |
| F3 | CGTTGAAACCAATCGTTGAGAA | |
| B3 | GACATCCTCGGTGGTGTG | |
| CPF | RGCYTTCACTGAAATGATGG | 53ºC |
| CPR | AACTGGAAGAATTGGCGGCCGCAGGAAT TTTTTTTTTTTTTTTTT |
Zróżnicowanie genetyczne
Analiza filogenetyczna przeprowadzona na podstawie genu kodującego białko płaszcza wykazała duże zróżnicowanie genetyczne wirusa i obecność czterech różnych szczepów w populacji wirusa: PVYNN242, PVYNWi-P, PVY0 i PVYNTN. Izolaty należące do PVYNTN były rekombinantami i zostały usunięte z drzewa filogenetycznego (Fig. 3).
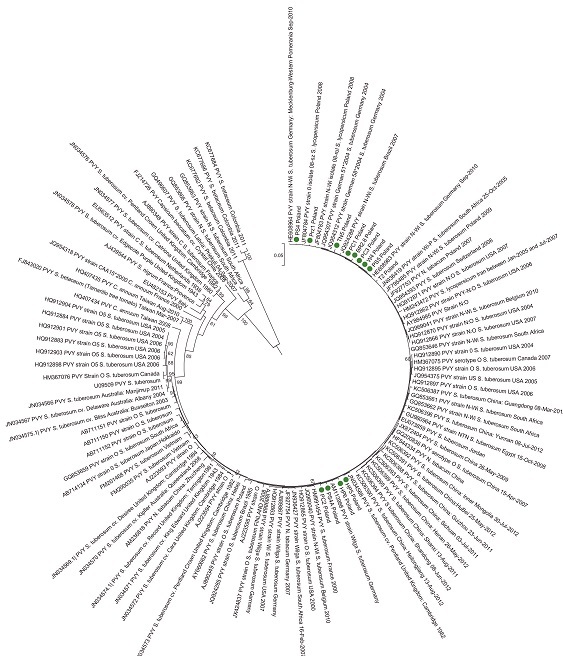
Fig. 3. Drzewo filogenetyczne utworzone w programie MEGA6 z wykorzystaniem sekwencji genu kodującego białko płaszcza polskich izolatów PVY i innych, których sekwencje zdeponowano w Banku Genów. Polskie izolaty oznaczono na zielono.
Literatura
1. Hasiów-Jaroszewska B, Stachecka J, Minicka J, Sowiński M, Borodynko N. 2015. Variability of Potato virus Y in tomato crops in Poland and development of a reverse transcription loop-mediated isothermal amplification method for virus detection. Phytopathology 105(9): 1270-1276
2. Rigotti S., Gugerli P. 2007. Rapid identification of potato virus Y strains by one-step triplex RT-PCR. Journal of Virological Methods 140: 90-94.