Clavibacter michiganensis subsp. michiganensis - Bakteryjny rak pomidora
Bakteryjny rak pomidora wywoływany przez bakterie Clavibacter michiganensis subsp. michiganensis jest chorobą o dużym znaczeniu ekonomicznym, w warunkach polskich występującą głównie w uprawach szklarniowych. Straty powodowane przez raka bakteryjnego szacuje się na 50-80%. Clavibacter michiganensis subsp. michiganensis (Cmm) znajduje się na liście obiektów kwarantannowych EPPO. W roku 1910 Smith po raz pierwszy opisał bakteryjne więdnięcie pomidora (Lycopersicon esculentum Mill.). Chorobę w szybkim tempie rozwijającą się w stanie Michigan w USA Smith nazwał „Grand Rapids disease”, a wywołującą ją bakterię „Bacterium michiganense”. Nazwa bakterii na przestrzeni lat zmieniała się od Pseudomonas michiganensis (Stevens, 1913), poprzez całkowitą zmianę grupy: Aplanobacter michiganense (Smih 1914), Phytomonas michiganense (Bergey i in., 1923), Mycobacterium flavum subsp. michiganense (Krasil’nikov, 1945) do Corynebacterium michiganense (Jensen, 1934). W roku 1986 Davis wydzielił z rodzaju Corynebacterium patogeny roślin i nadał im do dziś obowiązującą odrębną nazwę rodzajową Clavibacter.
Morfologia Komórki Cmm to proste lub nieznacznie zakrzywione gram-dodatnie pałeczki (Fig.1), wielkości 0,4-0,75x 0,8-2,5 µm, nieregularne często kształtu maczugowatego; przeważnie ułożone pojedynczo, ale często w parach kształt litery V, lub palisadowo. Bakterie ze starszych kultur przybierają niekiedy formę kokoidalną. Komórki są nieruchliwe, nie tworzą przetrwalników. Na pożywce SCM po siedmiu dniach wzrostu przybierają formę wypukłych, cylindrycznych koloni o barwie od jasnożółtej do mlecznożółtej o średnicy 3 – 8 mm.
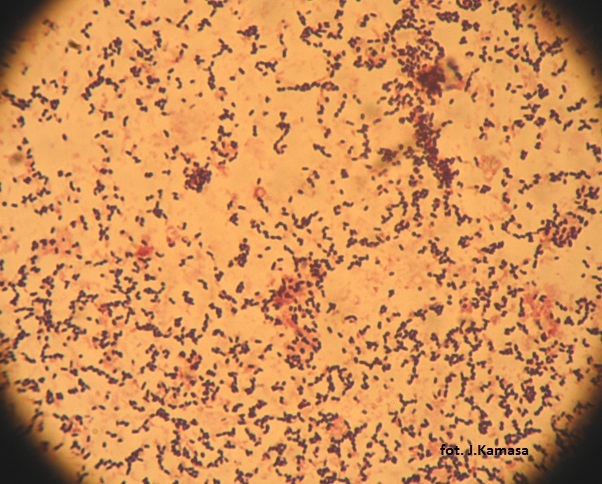
Fig. 1 Bakterie Cmm obecnow soku z łodygi pomidora w mikroskopie świetlnym, wybarwione metodą Grama wg Reed
Fizjologia Bakterie z rodzaju Clavibacter są obligatoryjnymi tlenowcami, wymagającymi pożywek bogatych w substancje odżywcze. Clavibacter michiganensis subsp. michiganensis zdolny jest do produkcji α-mannozidazy, β-ksylozidazy, endo-β-1,4-glukanazy/celulazy.Otymalna temperatura dla wzrostu bakterii z rodzaju Clavibacter wynosi od 25˚C do 28˚C. Większość bakterii posiada od 5 do 25 różnych kwasów tłuszczowych. Gatunki bakterii gram-dodatnich zwykle nie posiadają hydroksykwasów, ale bogate są w kwasy rozgałęzione. Względne ilości poszczególnych kwasów pozwalają dokładnie zróżnicować gatunek, podgatunek, a nawet biowar lub patowar. U szczepów Cmm pochodzących z różnych obszarów geograficznych stwierdzono obecność kwasu izopentakaprynowego (i15:0), anteizopentakaprynowego (a15:0), izopalmitynowego (i16:0) i anteizoheksakapryowego (a17:0). Chromosom Cmm składa się z 3,2 x 106 par zasad. Przeciętna zawartość guaniny i cytozyny w DNA jest wysoka, wynosi około 72% (Vidaver i Mandel 1974, Sasaki i in 1998, Engemann 2001). W komórce Cmm mogą znajdować się dwa plazmidy: pCM1 27,5-kb oraz pCM2 72-kb. Plazmid pCM1 zawiera gen celA kodujący wytwarzanie endo-β-1,4-glukanazy. Każdy izolat posiada w małej ilości kopii przynajmniej jeden z tych plazmidów. W plazmidzie pCM2 zlokalizowany jest gen pat-1, odpowiadający za wirulencję bakterii.
Objawy chorobowe Okres inkubacji dla bakteryjnego raka pomidora waha się od 7 dni do 4 tygodni, a natężenie objawów zależy od wieku i nawożenia rośliny, warunków środowiska, wielkości inokulum. Do typowych objawów bakteryjnego raka pomidora należy jednostronne więdnięcie liści, spowodowane zaczopowaniem wiązek przewodzących przez namnażające się bakterie. Więdnące liście brunatnieją i zamierają. Na łodygach, ogonkach liściowych i szypułkach kwiatowych pojawiają się ciemne smugi. Na silnie porażonych łodygach można zaobserwować pęknięcia, rakowate rany i śluzowate wycieki. Na łodygach przekrojonych wzdłuż często są widoczne żółtobrązowe smugi w miejscu wiązek przewodzących. W przypadku gwałtownie rozwijającej się infekcji obserwowane jest szybkie więdnięcie całego pędu rośliny. Znacznie rzadziej można zaobserwować objawy na owocach. Są to tak zwane „ptasie oczka” – małe ciemne plamy otoczone jasną obwódką o średnicy ok. 2 mm (Fig. 6-8).
Zakres roślin żywicielskich Typowym gospodarzem dla Cmm jest pomidor Lycopersicon esculentum. Istnieją doniesienia o naturalnych porażeniach innych roślin psiankowatych: Solanum nigrum S. Pimpinellifoli, S.trifolium. W wyniku sztucznej inokulacji Cmm poraża również Cyphomandra betacea, Nicotiana glutinosa, S. ammosum, S. muricatum, S. nigrum var. guneense).W warunkach laboratoryjnych podatne na infekcje są również S.melongena (oberżyna) i Capsicum annum (papryka).
Diagnostyka Metody molekularne oparte na technice PCR- zalecane jest stosowanie starterów zaprojektowanych przez Pastric K.H., Rainey F.A.(1999). W przypadku izolatów wirulentnych zastosowanie znalazły startery zaprojektowane przez Dreier i in. (1994) amplifikujące fragment DNA o długości 614 bp leżący w obrębie genu pat-1. System Identyfikacji bakterii BIOLOG –system polega na analizie profilu biochemicznego izolatów i z powodzeniem pozwala na identyfikację bakterii Cmm oraz odróżnienie ich od innych podgatunków, należących do rodzaju Clavibacter. W przypadku wykrywania bakterii Cmm na powierzchni nasion obowiązują protokoły EPPO (OEPP/EPPO Bulletin. 1992 . Quarantine procedure: Clavibacter michiganensis ssp . michiganensis - Test methods for tomato seeds. 22, 219-224)i ISTA (Handbook on Seed Health Testing Working Sheet No 67).
Bakteryjny rak pomidora od wielu lat jest obiektem badań w Instytucie Ochrony Roślin, a obecnie w Klinice Chorób Roślin. W ramach badań prowadzonych w latach 1999 -2004 oszacowano zróżnicowanie populacji izolatów Cmm zebranych na terenie Polski, głownie w okolicach Kalisza.Jedną z zastosowanych była metoda RFLP (Restriction Fragments Length Polymorphism – polimorfizm długości fragmentów restrykcyjnych).Powielony przy użyciu starterów CBR16F2 i CBR16R2 fragment DNA wielkości 1,3 tys. pz poddano analizie RFLP wykorzystując enzymy restrykcyjne EcoRI, BamHI, Hind, Ssp, Dra, EcoRI oraz BamHI. Produkty trawienia rozdzielane były na 1% żelu poliakrylamidowym i wybarwiane bromkiem etydyny (Fig. 2).
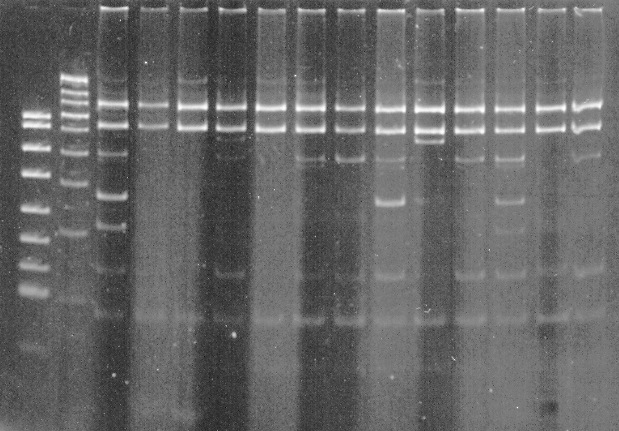 Fig.2 Obraz elektroforetyczny produktów trawienia enzymem EcorI
Fig.2 Obraz elektroforetyczny produktów trawienia enzymem EcorI
Jako metodę klasyfikacji wybrano analizę skupień, grupowania dokonano metodą aglomeracji pełnego wiązania. Na podstawie analizy restrykcyjnej podzielono izolaty na trzy grupy. Odległość wiązań pomiędzy I i II wynosiła 2,6 a pomiędzy trzecią i dwiema pierwszymi 2,8. Najmniej liczna okazała się grupa III, w skład której weszło 10 izolatów. Wszystkie najsilniej patogeniczne izolaty znalazły się w grupie I i II, przy czym izolaty o numerach 72, 70, 69, 68, 83 porażające silnie 100% roślin znalazły się na diagramie w swoim najbliższym sąsiedztwie, a odległość wiązań pomiędzy nimi wynosiła 0 (Fig.3)
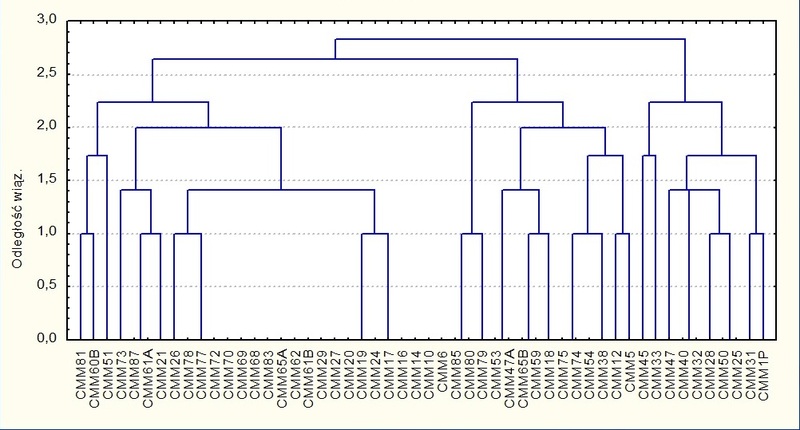
Fig.3 Dendogram przedstawiający zróznicowanie populacji Cmm na podstawie analizy RFLP
Drugą metodą zastosowaną do oceny zróżnicowania populacji Cmm był Rep-PCR (Repetitive extragenic palindromic-PCR). W wyniku reakcji Rep-PCR ze starterami ERIC otrzymano 16 cech, ze starterem BOX 15 cech, ze starterami REP 10 cech (Fig.4).
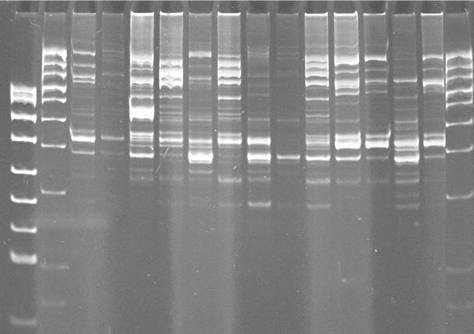 Fig.4 rep-PCR ze starterami BOX
Fig.4 rep-PCR ze starterami BOX
Niektóre izolaty miały identyczne wzory prążków, więc połączono je w grupy. W przypadku starterów ERIC uzyskano 52 grupy, dla startera BOX 37 grup, a dla REP 20 grup. Do konstrukcji drzew zastosowano metodę UPGMA, współczynniki dystansu zostały obliczone przy użyciu algorytmu Sokal and Sneath 3. Diagramy mający postać promieniście rozgałęzionych drzew generowano w programie TreeView 32 (Fig.5), przyjmując za grupę zewnętrzną Clavibacter michiganensis subsp. insidiosus, oznaczony jako ci. W przypadku starterów ERIC zaobserwowano pewną zbieżność pomiędzy miejscem pochodzenia izolatu, a jego położeniem na diagramie w formie drzewa. Izolaty o numerach Cmm52, Cmm59, Cmm60 pochodzące ze Skaryszewa znalazły się w grupach oznaczonych kolorem zielonym, podobnie jak izolaty Cmm86, Cmm86a, Cmm87 pozyskane z upraw w okolicy Torunia w roku 2001 i izolaty Cmm77, Cmm78 wyizolowane w roku 2001 z roślin pochodzących z Bijewa. Grupy oznaczone na rycinie 2 kolorem pomarańczowym, najbardziej oddalone od centrum drzewa skupiają izolaty Cmm5, Cmm11, Cmm63, Cmm90. Dwa pierwsze izolaty charakteryzują się nieco innym wyglądem koloni bakteryjnej, są one jaśniejsze i wytwarzają bardzej gęsty i kleisty śluz niż pozostałe. Izolat Cmm11 jest słabo wirulentny i wymaga temperatury 27˚C do wywołania reakcji nadwrażliwości na Mirabilis jalapa, stanowiąc pod tym względem mniejszość w badanej populacji. W reakcji PCR ze starterem BOX najczęściej występujące prążki miały wielkość: 1000, 950, 650, 440, 380 par zasad. Izolaty Cmm86, Cmm86a, Cmm86b, Cmm87 pochodzące z okolic Torunia, wyizolowane w 2001 znalazły się w grupach oznaczonych jasnoniebieskim kolorem. Izolaty należące do grup c14 i c16 zaznaczone kolorem fioletowym nie różniły pod innymi względami od pozostałych, a ich miejsce na diagramie można wyjaśnić jakością matrycy DNA użytej w reakcji PCR. Jeżeli zastosowane w badaniu DNA było podegradowane, w profilu prążków mogło zabraknąć fragmentów o większej ilości par zasad. W reakcji PCR ze starterami REP najczęściej występujące prążki miały wielkość: 1031, 750 (wszystkie izolaty), 180, ok. 110 par zasad. Rzadziej występowały prążki o wielkości 550 i 270 pz. Nie zaobserwowano związku pomiędzy miejscem pochodzenia izolatów, a ich pozycją na diagramie. Izolaty pochodzące z upraw w okolicy Kalisza znalazły się we wszystkich grupach.
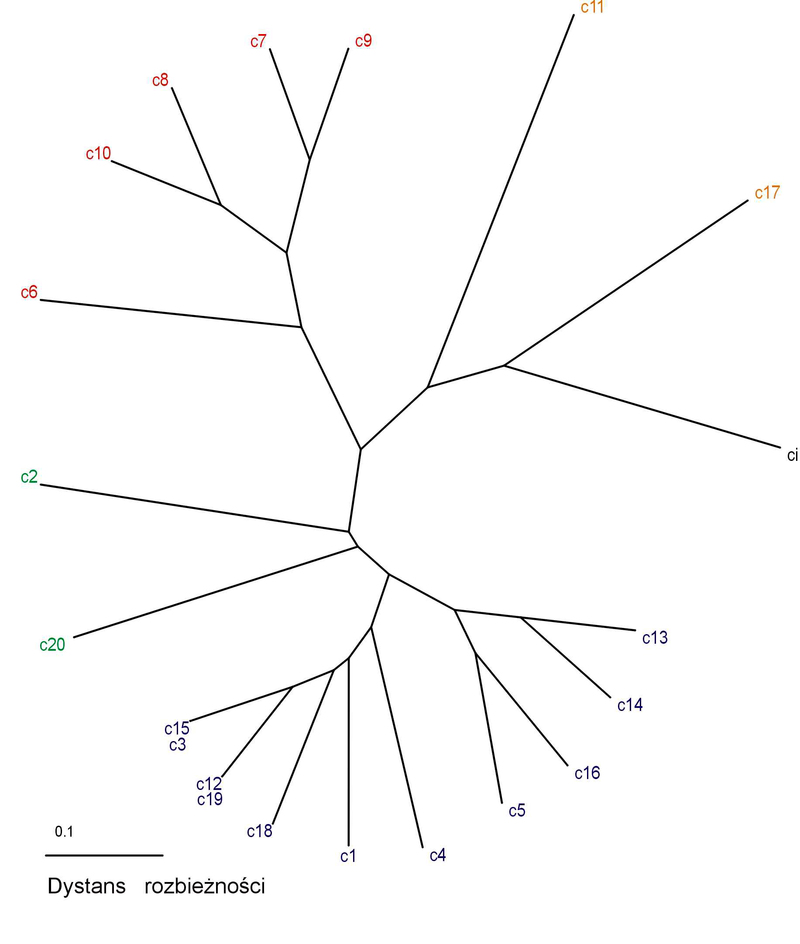
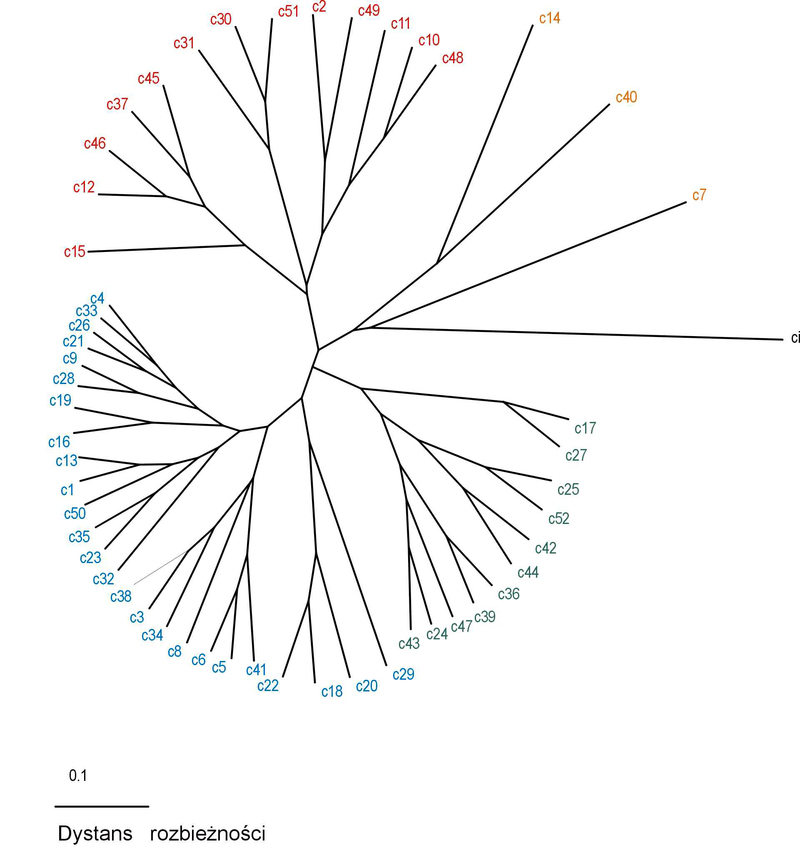
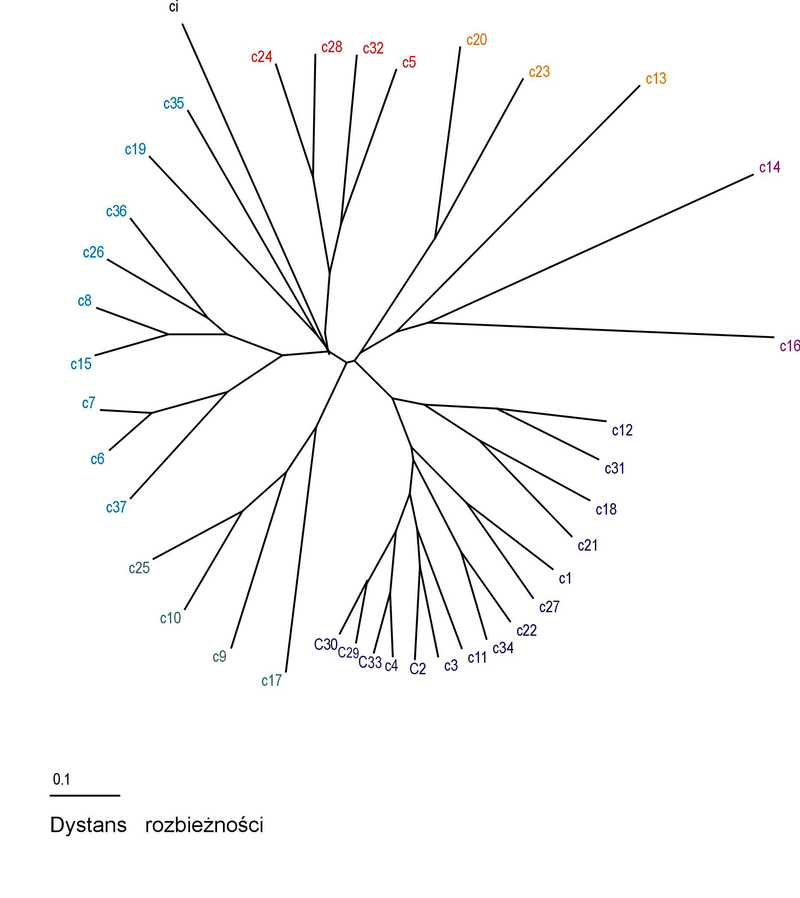
Fig. 5 Dendogramy przedstawiające zróżnicowanie populacji Cmm na podstawie rep-PCR
Fig. 6-8 Objawy bakteryjnego raka pomidora



"Ptasie oczka" na owocach przebarwienia wiązek w łodydze nekrozy na liściach
Startery do detekcji Clavibacter michiganensis ssp. michiganensis w reakcji PCR
|
starter |
wielkość produktu |
region |
referencje |
|
CMM-5: 5′- GCG AAT AAG CCC ATA TCA A -3′
CMM-6: 5′- CGT CAG GAG GTC GCT AAT A -3′ |
614 nt |
gen pat-1 na plazmidzie |
Dreier J, Bermpohl A & Eichenlaub R (1995) Southern hybridization and PCR for specific detection of phytopathogenic Clavibacter michiganensis subsp. michiganensis. Phytopathology 85, 462–468 |
|
PSA-4: 5′-TCA TTG GTC AAT TCT GTC TCC C -3′
PSA-R: 5′-TAC TGA GAT GTT TCA CTT CCC C -3′ |
270 nt |
16S–23S rRNA ITS |
Pastrik KH & Rainey FA (1999) Identification and differentiation of Clavibacter michiganensis subspecies by Polymerase Chain Reaction-based techniques. Journal of Phytopathology 147, 687–693 |