Pantoea ananatis - Plamistość liści kukurydzy (ang. Leaf spot disease)
Pantoea ananatis – plamistość liści kukurydzy (ang. Leaf spot disease)
Występowanie i zakres gospodarzy:
P. ananatis jest gatunkiem polifagicznym i występuje powszechnie. Część należących do tego gatunku szczepów jest epi – lub endofitami i jest niepatogeniczna dla roślin. Gatunek ten może być również oportunistycznym patogenem człowieka. P. ananatis jest patogenem kukurydzy, agawy, sorga, kantalupy, melona, cebuli, eukaliptusa, ryżu oraz grzybów (Pleurotus eryngii).
Rozprzestrzenianie:
Bakteria ta wnika do wnętrza roślin przez ich naturalne otwory (aparaty szparkowe i hydatody), niesione kroplami wody oraz przez mikrouszkodzenia roślin powstające najczęściej w czasie silnych i zacinających deszczy, i wiatrów niosących cząsteczki brudu i drobinki piasku, jak również przez większe uszkodzenia roślin powstałe, np. w czasie gradobicia. Jej obecność na roślinie kukurydzy nie jest równoznaczna z rozwojem choroby. Niektóre szczepy tego gatunku mogą występować na kukurydzy naturalnie i kolonizować powierzchnię jej liści i przestrzenie międzykomórkowe, nie powodując przy tym wystąpienia objawów.
Szkodliwość:
Paccola-Meirelles i wsp. (2001) opisali bakteryjnego patogena roślin kukurydzy, który powodował chorobę zwaną „Phaeosphaeria leaf spot”. Choroba ta powodowała duże szkody w uprawach kukurydzy w Brazylii w latach 90-tych i obecnie występuje tam na wszystkich obszarach, w których uprawiana jest kukurydza. W sprzyjających warunkach może ona powodować rozległe uszkodzenia liści oraz znaczne straty w plonie ziarna. Pierwotnie choroba ta została omyłkowo uznana za grzybową, powodowaną przez gatunek Phaeosphaeria maydis (syn. Sphaerulinia maydis). Późniejsze badania wykazały jednak że prawdziwym czynnikiem sprawczym tej choroby jest bakteria P. ananatis . Bakteria ta powoduje również inną chorobę kukurydzy zwaną bakteryjnym gniciem łodygi (ang. bacterial stalk rot). Objawami charakterystycznymi dla tej choroby są karłowacenie i występowanie pionowego pęknięcia w łodydze na wysokości pierwszego międzywęźla. W wielu zainfekowanych roślinach pęknięcie było obecne również na drugim i na trzecim międzywęźle. Po otwarciu międzywęźla widoczne były zmiany chorobowe wzdłuż miejsca pęknięcia oraz wewnętrzne brązowienie tkanki łodygi rozszerzające się w górę rośliny w niektórych przypadkach sięgające szczytu rośliny. Kolby były niedorozwinięte. Czasami obserwuje się mięknięcie, więdnięcie brzegów liści, blaknięcie ich i zwijanie do wewnątrz.

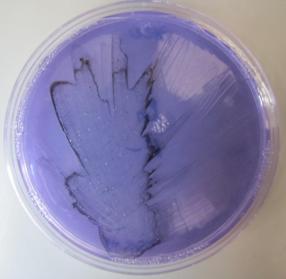
Fig. 1. Objawy chorobowe na kukurydzy wywołane przez Pantoea ananatis

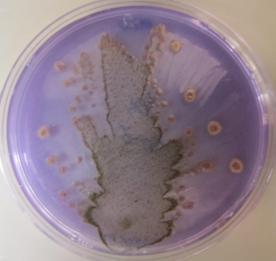
Fig. 2. Objawy chorobowe na kukurydzy wywołane przez Pantoea ananatis
Wykrywanie i identyfikacja
Do wykrywania P. ananatis stosowaliśmy szereg technik badawczychnależących do metody biochemicznej: wykrywanie na pożywce półselektywnej PA20, identyfikacja przy pomocy testów API 20E, BIOLOG GEN II i GEN III, oraz metody molekularnej klasyczny PCR z wykorzystaniem starterów specyficznych, Bio-PCR, Real-Time PCR, HRM-PCR i MLSA.
Morfologia i charakterystyka biochemiczna:
Rodzaje Pantoea tworzą pojedyncze komórki bakterii zaliczane do pałeczek prostych o wymiarach od 1,0 do 1,0-3,0 µm. Mają wiele rzęsek rozmieszczonych perytrychalnie. Jedynym wyjątkiem jest gatunek P. stewartii, który nie posiada rzęsek i jest nieruchliwy. Komórki tych bakterii występują pojedynczo, parami lub w formie krótkich łańcuszków. Nie tworzą przetrwalników. Pantoea ananatis jest bakterią fakultatywnie beztlenową. Na podłożu YDC jej kolonie są żółte i wypukłe, mogą być również wodniste i ciągnące się. Wykazują one charakterystykę typową dla rodzaju Pantoea. Z bakterią tą często mylone są szczepy blisko spokrewnionego z nią gatunku P. agglomerans (syn. E. herbicola). Do selektywnego wykrywania P. ananatis z tkanki roślinnej opracowano selektywne podłoże PA 20 (Fig. 3 i 4.).
 A
A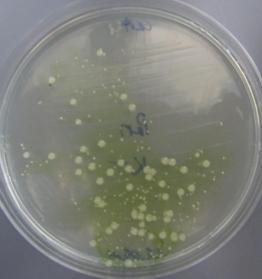 B
B
Fig. 3. Sok roślinny pochodzący ze zdrowej rośliny kukurydzy naniesiony równolegle na pożywki PA20 (A) i TSA (B).
 A
A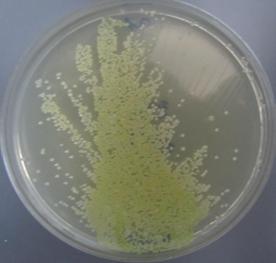 B
B
Fig. 4. Sok roślinny ze zdrowej kukurydzy z dodatkiem wodnej zawiesiny bakteryjnej izolatu z kolekcji własnej M304 naniesiony równolegle na pożywki PA20 (A) oraz TSA (B).
W teście API 20E badane izolaty P. anantis zostały zidentyfikowane na podstawie 7-cyfrowego kodu charakterystycznego dla tego gatunku. Na przykładzie izolatu M408 był to kod: 1205573.
Do identyfikacji P. ananatis wykorzystano również system Biolog. Przy pomocy tego systemu izolaty: M241, M304, M408 i M471 zidentyfikowano, jako należące do gatunku P. ananatis. Wynik ten w postaci dendrogramu przedstawia rysunek 5.
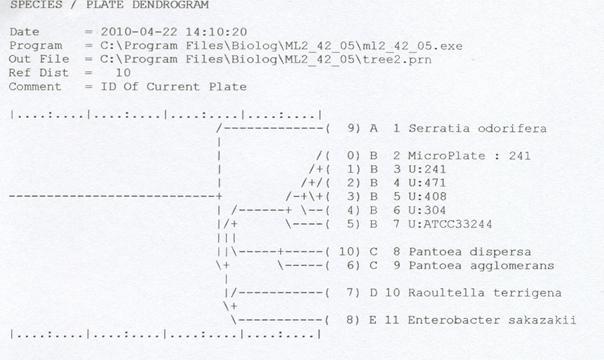
Fig. 5. Dendrogram przedstawiający identyfikację i porównanie izolatów P. ananatis do innych izolatów dostępnych w bazie danych systemu BIOLOG.
Metoda molekularna:
Do idetyfikacji P. anantis wykorzystano również klasyczną reakcję PCR ze starterami specyficznymi dla P. ananatis. Spośród 3 testowanych par starterów, jedynie para PanITS1/EC5-Gi wykrywała wszystkie izolaty tego gatunku użyte w tym doświadczeniu (M241, M304, M408, M471, Pan868 i ATCC 33244). Za pomocą pary starterów: PanITS1 / EC5 nie udało się wykryć izolatu M471, natomiast para Pan16S/Pan16AS nie pozwalała na wykrycie obecności referencyjnego izolatu P. ananatis ATCC 33244.
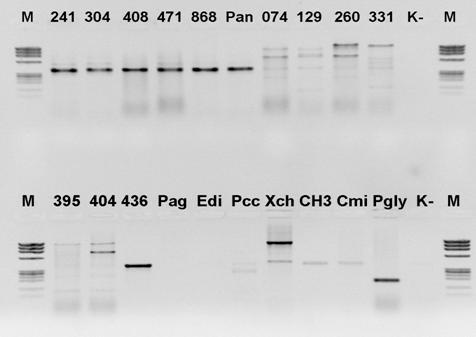
Fig. 6. Detekcja P. ananatis przy użyciu pary starterów: PanITS1 / EC5-Gi. Górny rząd od lewej: 241, 304, 408, 471, 868 i Pan – izolaty P. ananatis; 074, 129, 260, 331, 395, 404, 436 i Pag – izolaty P. agglomerans; Edi-E. cloaceae subsp. dissolvens, Xch-X. campestris pv. chrysanthemii, Cmi-C. michiganensis subsp. insidiosus, Pgly-P. syringae pv. glicynea; M – marker DNA ØX174, K- kontrola ujemna.
Zbadano również czułość reakcji PCR z użyciem pary starterów PanITS1 / EC5-Gi i określono ją na 0,5 pg DNA bakteryjnego / mikrolitr. Wyniki doświadczenia przedstawiono na rysunku nr 7B.
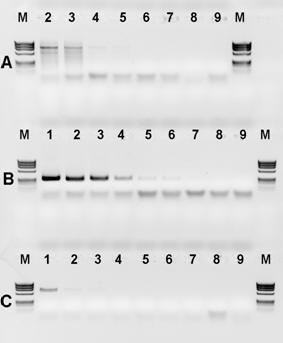 1
1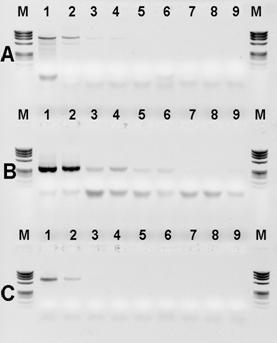 2
2
Fig. 7. Czułość detekcji genomowego DNA P. ananatis. Rząd A: startery PanITS1/EC5, rząd B: startery: PanITS1/EC5-Gi, rząd C: startery: Pan16S/Pan16AS. Dla wszystkich rzędów-od lewej: linia 1: 50 ng, linia 2: rozcieńczenie 10-1 (5 ng), linia 3: rozcieńczenie 10-2 (0,5 ng), linia 4: rozcieńczenie 10-3 (50 pg), linia 5: rozcieńczenie 10-4 (5 pg), linia 6: rozcieńczenie 10-5 (0,5 pg), linia 7: rozcieńczenie 10-6 (0,05 pg), linia 8: rozcieńczenie 10-7 (0,005 pg), linia 9: kontrola wodna. Marker DNA (M): ØX174; 1 – izolat ATCC 33244, 2 – izolat M304.
Bio-PCR
Do podniesienia czułości detekcji P. ananatis w klasycznej reakcji PCR, zastosowano również technikę Bio-PCR. Jest to odmiana techniki PCR w której czułość standardowej reakcji PCR wzbogacona została o etap wstępnej hodowli bakterii. Polega to na wysianiu soku roślinnego z potencjalnie zakażonej rośliny, na podłoże mikrobiologiczne i jego 24 godzinnej hodowli celem podniesienia liczebności komórek bakteryjnych. Po zakończeniu inkubacji wszystkie wyrosłe na podłożu mikroorganizmy są z niego zmywane i poprzez gotowanie przygotowywana jest z nich matryca DNA do reakcji PCR. Dzięki temu etapowi komórki patogena występujące w roślinie nielicznie i nie wywołujące jeszcze symptomów chorobowych, mogą zostać namnożone, a przez to ich obecność zostanie wykazana w reakcji PCR. Dlatego Bio-PCR jest doskonałym uzupełnieniem konwencjonalnej reakcji PCR.
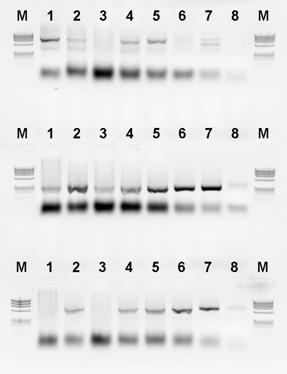
Para starterów PanITS1/EC5-Gi pozwoliła na wykrycie izolatów P. ananatis koncentracji 10-11, co w naszym doświadczeniu odpowiadało koncentracji 3 jtk/ml (Fig. 8).
 A
A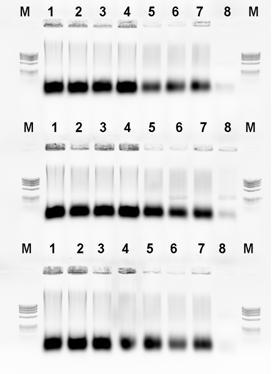 B
B
Fig. 8. Wynik detekcji P. ananatis przy pomocy techniki Bio-PCR. Rząd 1 od góry: startery PanITS1/EC5, rząd 2: startery PanITS1/EC5-Gi, rząd 3: startery Pan16S/Pan16AS. Dla wszystkich rzędów-od lewej: linia 1: 10-5, linia 2 do 6: kolejne rozcieńczenia dziesiętne, linia 7: rozcieńczenie 10-11, linia 8: kontrola wodna. M: Marker DNA ØX174. A – izolat ATCC 33244, B – izolat M304.
Dodatkowo przeprowadzono również analizę filogenetyczną wybranych izolatów bakteryjnych należących do gatunków: P. ananatis, w oparciu o sekwencję genu 16S rRNA (ok. 1500 pz). Do amplifikacji tego fragmentu genomu, używano równolegle 3 par starterów (Fig. 9). Otrzymane produkty PCR oczyszczono i poddano sekwencjonowaniu.
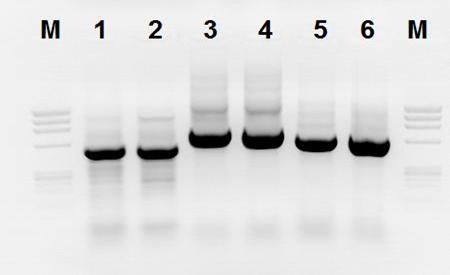
Fig. 9. Produkty PCR trzech par starterów służących do namnażania 16S rDNA. Oznaczenia: Od lewej: linia 1 - marker DNA ØX174, linie 2 i 3 – produkt PCR pary starterów 16S01/pD2, linie 4 i 5 – produkt PCR pary starterów 16S02/pF2, linie 6 i 7 - produkt PCR pary starterów 16S04/pH2.
Bazując na uzyskanych sekwencjach nukleotydowych genu 16S rRNA wybranych izolatów oraz na analogicznych do nich sekwencjach pobranych z GenBanku, przeprowadzono analizę filogenetyczną (Fig. 10). Analizę przeprowadzono przy użyciu metody najbliższego sąsiada (ang. neighbor joining) w programie Mega 4.0. Jako wartość bootstrap przyjęto 1000 powtórzeń. Dane umożliwiające identyfikację izolatów są umieszczone na dendrogramie (Fig. 10). Sekwencje genu 16S rRNA, uzyskane dla wybranych izolatów, po analizie bioinformatycznej zamieszczono w bazie danych GenBank (Tab. 1).
Tab. 1. Uzyskane sekwencje genu 16S rDNA zamieszczone w bazie danych GenBank.
|
LP. |
Izolat |
Długość sekwencji nukleotydowej (pz) |
Numer akcesyjny GenBanku |
Gatunek |
|
1. |
M241 |
1477 |
GQ332197 |
P. ananatis |
|
2. |
M304 |
1508 |
GU565633 |
P. ananatis |
|
3. |
M408 |
1508 |
GQ332198 |
P. ananatis |
|
4. |
M471 |
1464 |
GU324264 |
P. ananatis |
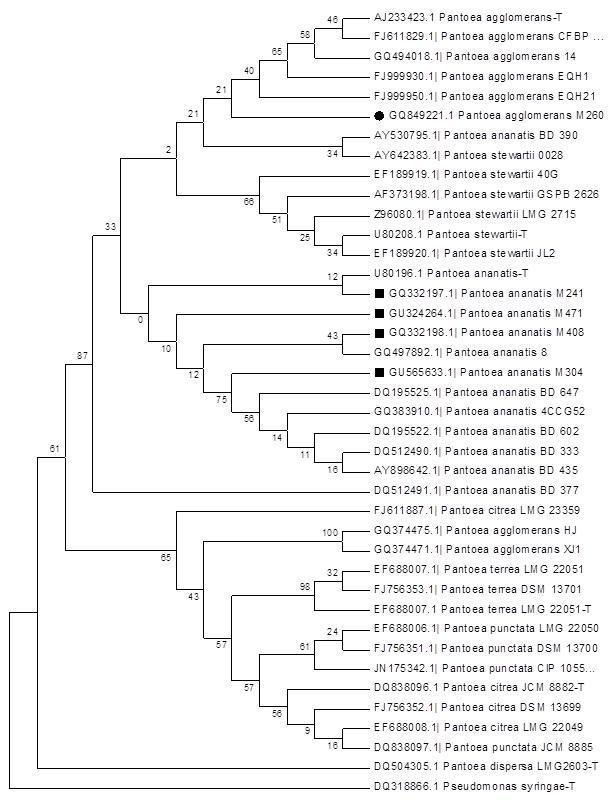
Fig. 10. Dendrogram przedstawiający analizę sekwencji genu 16S rRNA badanych izolatów zidentyfikowanych jako P. ananatis i P. agglomerans, skonstruowany metodą neighbor joining. W dendrogramie uwzględniono różne gatunki rodzaju Pantoea oraz 1 sekwencję Pseudomonas syringae (AF094749) użytą, jako grupa zewnętrzna. Wartość bootstrap wynosiła 1000 powtórzeń. Badane izolaty P. ananatis oznaczono na dendrogramie czarnymi kwadratami, natomiast badany izolat P. agglomerans oznaczono czarnym kołem. Sekwencje szczepów wzorcowych (ang. type strains) każdego analizowanego gatunku oznaczone są literą: t (-T), za nazwą gatunkową.
Na przykładzie P. ananatis przeprowadzono ocenę przydatności techniki Real-Time PCR do identyfikacji i masowej detekcji bakterii występujących na kukurydzy. Jest to technika czuła, specyficzna i stosunkowo szybka. Zaprojektowano własne startery do detekcji P. ananatis i zoptymalizowano warunki przeprowadzenia reakcji Real-Time PCR. Zaprojektowane startery PanITS1/S01R amplifikowały fragment genu 16S rDNA. Elektroforetyczny rozdział mieszaniny poreakcyjnej Real-Time PCR wykazał obecność tylko specyficznych dla P. ananatis produktów reakcji (Fig. 11).
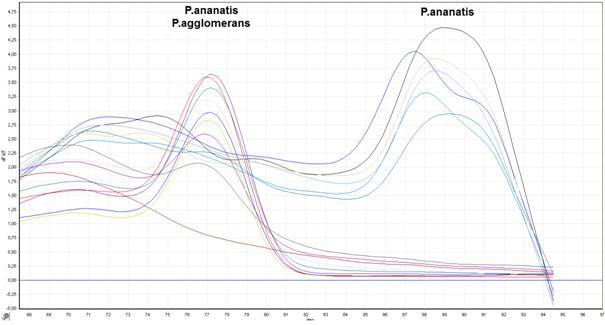
Fig. 11. Przebieg krzywych dysocjacyjnych (krzywych topnienia) produktów reakcji Real-Time PCR: Izolaty: P. ananatis: M241, M304, M408, M471, Pan 868, ATCC 33244; P. agglomerans: M074, M129, M260, M331, M395, ATCC 33243. Kontrola negatywna: kolor brązowy.
Na potrzeby detekcji P. anantis i odróżnienie jej od blisko z nią spokrewnionej P. agglomerans zoptymalizowano reakcję HRM-PCR (PCR połączony z wysokorozdzielczą analizą denaturacji), w oparciu o region zmienny 16S-23S ITS rRNA. Technika ta umożliwiła genetyczne rozróżnienie tych dwóch gatunków bakterii. Uzyskane wyniki wskazują, że zaprojektowane do HRM-PCR startery specyficznie amplifikują analizowany fragment genomu P. ananatis. Analiza krzywej topnienia produktów reakcji wykazała obecność pojedynczego produktu amplifikacji dla każdej z przeprowadzonych reakcji (Fig. 12).
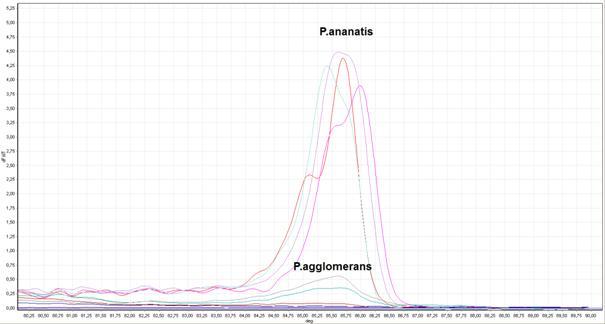
Fig. 12. Krzywe topnienia produktów reakcji HRM-PCR. Izolaty: P. ananatis: M241, M471, Pan868, ATCC 33244; P. agglomerans: M074, M129, M260, M331, M395. Kontrola ujemna – kolor brązowy.
Dla wybranych wcześniej badanych izolatów P. ananatis i P. agglomernas, przeprowadzono również analizę MLSA. Technikę tą zastosowano do badania zamplifikowanych i zsekwencjonowanych czterech genów: atpD, fusA rplB i rpoB, uzyskanych dla badanych izolatów P. ananatis i P. agglomerans. Sekwencje te poddano analizie programem DNAsp v5.0, obliczając stopień różnorodności i poziom polimorfizmu badanych sekwencji. Stopień zróznicowania nukleotydów różnił się pomiędzy badanymi genami. Zakres liczby miejsc polimorficznych wynosił od 51 dla genu rplB do 279 dla połączonych sekwencji wszystkich czterech badanych genów. Natomiast średnia liczba różnic nukleotydowych na miejsce (pi) wynosiła od 3% dla genu rplB do 39,6% dla połączonych sekwencji badanych genów. Wyniki analizy przedstawiono na rysunku 11. Technika MLSA potwierdziła prawidłowość działania wcześniej stosowanych technik identyfikacji.
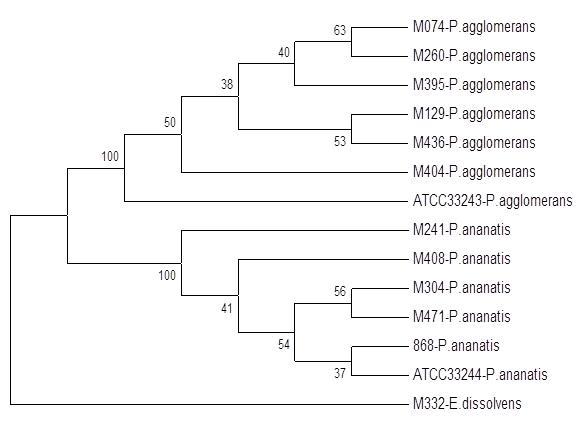
Fig. 13. Dendrogram przedstawiający analizę sekwencji genów: atpD, fusA rplB i rpoB, skonstruowany metodą maksymalnego podobieństwa (ang. maximum likelihood) 13 izolatów z rodzaju Pantoea i jednego z rodzaju Enterobacter. Numery izolatów odpowiadają tym zebranym w tabeli 19. M332 - E. cloaceae subsp. dissolvens (grupa zewnętrzna); ATCC – izolaty referencyjne z kolekcji ATCC.
Chwasty polne jako potencjalny rezerwuar bakterii patogenicznych dla kukurydzy
Problem naturalnych rezerwuarów bakterii fitopatogenicznych został w literaturze naukowej opisany w odniesieniu do chwastów występujących w uprawach i spełniajacych czasami rolę rezerwuaru bakterii fitopatogenicznych w warunkach naturalnych. Zagadnienie to zbadano również dla P. anantis. W wyniku sztucznej inokulacji pospolitych gatunków chwastów bakterią P. anantis, w warunkach szklarniowych, objawy infekcji bakteryjnej wykazały dwa gatunki chwastów: chwastnica jednorodna i palusznik krwawy (izolat M408). Bakterie reizolowane z roślin wykazujących objawy infekcji bakteryjnej, wykazywały identyczny profil biochemiczny w testach API 20E, jak izolaty, których użyto do zakażania, co jednoznacznie potwierdziło ich tożsamość. Uzyskane wyniki pozwalają na założenie, że w sprzyjających warunkach środowiskowych wybrane gatunki pospolicie występujących chwastów polnych mogą stanowić potencjalne siedlisko bakterii patogenicznych dla kukurydzy.
Tab. 2. Wyniki sztucznej inokulacji roślin pospolitych chwastów polnych izolatami bakteryjnymi patogenicznymi dla roślin kukurydzy
|
Roślina |
Izolat numer |
|||||||||
|
074 |
241 |
260 |
336 |
391 |
393 |
408 |
425 |
436 |
722 |
|
|
Chwastnica jednostronna |
- |
- |
- |
- |
- |
- |
+ |
+ |
- |
+ |
|
Perz właściwy |
- |
- |
- |
- |
- |
- |
- |
- |
- |
- |
|
Wiechlina roczna |
- |
- |
- |
- |
- |
- |
- |
- |
- |
- |
|
Wyczyniec polny |
- |
- |
- |
- |
- |
- |
- |
- |
- |
- |
|
Włośnica zielona |
- |
- |
- |
- |
- |
- |
- |
- |
- |
- |
|
Palusznik krwawy |
- |
- |
- |
- |
- |
- |
+ |
- |
- |
- |
|
Życica trwała |
- |
- |
- |
- |
- |
- |
- |
- |
- |
- |
|
Życica wielokwiatowa |
- |
- |
- |
- |
- |
- |
- |
- |
- |
- |
|
Miotła zbożowa |
- |
- |
- |
- |
- |
- |
- |
- |
- |
- |
|
Owies głuchy |
- |
- |
- |
- |
- |
- |
- |
- |
- |
- |
+ - wystąpienie objawów chorobowych; - - brak objawów chorobowych.
DDodatek:
Tab. 1. Oligonukleotydy użyte w trakcie badań do namnażania genu 16S rDNA oraz w metodzie FAFLP.
|
Starter |
Region amplifikowany |
Sekwencja 5’à 3’` |
Ta (ºC) |
Długość produktu PCR (pz) |
Źródło |
Zastosowanie w badaniach |
|
U1F |
16S rDNA |
CTYAAAKRAATTGRCGGRRRSSC |
55 |
495 |
Rivas et al. 2004 |
Namnażanie 16S rDNA |
|
U1R |
16S rDNA |
CGGGCGGTGTGTRCAARRSSC |
55 |
495 |
Rivas et al. 2004 |
Namnażanie 16S rDNA |
|
FGPS1490 |
16S-23 S ITS rDNA |
TGCGGCTGGATCACCTCCTT |
60 |
630 |
Parker 2002 |
Namnażanie 16S rDNA |
|
FGPL132 |
16S-23S ITS rDNA |
CCGGGTTTCCCCATTCGG |
60 |
630 |
Parker 2002 |
Namnażanie 16S rDNA |
|
16S01 |
16S rDNA |
AGTTTGATCCTGGCTCAG |
57 |
491 |
Gerischer i wsp. 2008 |
Namnażanie 16S rDNA |
|
pD2 |
16S rDNA |
GTA TTA CCG CGG CTG CTG |
57 |
491 |
Edwards i wsp. 1989 |
Namnażanie 16S rDNA |
|
16S02 |
16S rDNA |
CTCCTACGGGAGGCAGCAGT |
57 |
695 |
Gerischer i wsp. 2008 |
Namnażanie 16S rDNA |
|
pF2 |
16S rDNA |
ACG AGC TGA CGA CAG CCA TG |
57 |
695 |
Edwards i wsp. 1989 |
Namnażanie 16S rDNA |
|
16S04 |
16S rDNA |
AACTCAAAGGAATTGACGG |
63 |
596 |
Gerischer i wsp. 2008 |
Namnażanie 16S rDNA |
|
pH2 |
16S rDNA |
AAG GAG GTG ATC CAG CCG CA |
63 |
596 |
Edwards i wsp. 1989 |
Namnażanie 16S rDNA |
|
EcoRI-1 |
- |
CTCGTAGACTGCGTACC |
- |
- |
Vos i wsp. 1995 |
FAFLP Adaptor EcoRI |
|
EcoRI-2 |
- |
CATCTGACGCATGGTTAA |
- |
- |
Vos i wsp. 1995 |
FAFLP Adaptor EcoRI |
|
MseI-1 |
- |
GACGATGAGTCCTGAG |
- |
- |
Vos i wsp. 1995 |
FAFLP-Adaptor MseI |
|
MseI-2 |
- |
TACTCAGGACTCAT |
- |
- |
Vos i wsp. 1995 |
FAFLP-Adaptor MseI |
|
Eco-00 |
fragmenty genomu |
GACTGCGTACCAATTC |
56 |
- |
Brady i wsp. 2007 |
FAFLP |
|
Mse-00 |
fragmenty genomu |
GATGAGTCCTGACTAA |
56 |
- |
Brady i wsp. 2007 |
FAFLP |
|
Eco-C |
fragmenty genomu |
GACTGCGTACCAATTCC |
66-56 |
- |
Brady i wsp. 2007 |
FAFLP |
|
Mse-GC |
fragmenty genomu |
GATGAGTCCTGAGTAAGC |
66-56 |
- |
Brady i wsp. 2007 |
FAFLP |