Ca. Phytoplasma asteris - Choroba fitoplazmatyczna pomidora
Candidatus Phytoplasma asteris - Fitoplazmatyczna choroba pomidora.
Wstęp
Występowanie i zakres gospodarzy
Pomidor (Solanum lycopersicum) jest jedną z najważniejszych ekonomicznie upraw w wielu krajach Europy. Jego produkcja jest często ograniczona przez skutki działania wielu szkodników owadzich i chorób przez nie przenoszonych, wliczając w to choroby fitoplazmatyczne (Del Serrone i wsp. 2001). Fitoplazmy infekują kilkaset gatunków roślin takich jak warzywa, owoce, rośliny ozdobne i drzewa (Lee i wsp. 2000). Występują w wielu różnych środowiskach, od klimatu umiarkowanego do tropikalnego (Arocha i wsp. 2005, Palmano 2001). Szkody upraw powodowane przez choroby fitoplazmatyczne mają istotny i szkodliwy wpływ na rolnictwo (Del Serrone et al. 2001). Fitoplazmy są patogenami systemicznymi. Pierwotnie opisywano je jako należące do mykoplazm (ang. mycoplasma-like organisms (MLOs)) (Palmano 2001). Są to bakterie nie posiadające ściany komórkowej i należące do klasy Mollicutes (Arocha i wsp. 2005; Bertaccini i wsp. 2005; Palmano 2001). Przeważnie są mniej lub bardziej okrągłego kształtu, ze średnicą 200 – 300 nm (Al-Zadjali i wsp. 2007). Każda komórka fitoplazmy otoczona jest przez trójwarstwową błonę komórkową o grubości 8 – 10 nm (Lee i wsp. 2001). Ich strukturę wewnętrzną tworzą rybosomy i łańcuchy DNA (Al-Zadjali i wsp. 2007). Patogeniczne dla roślin fitoplazmy zamieszkują i pasożytują w tkance łyka rośliny gospodarza (Lee i wsp. 2000; Palmano 2001). Są przenoszone przez żywiące się łykiem owady, głównie skoczki (Cicadellidae), czasem miodówki (Psyllidae) (Arocha i wsp. 2005; Lee i wsp. 2000). Fitoplazmy mogą powodować zaburzenia w równowadze hormonów roślinnych i regulatorów wzrostu w roślinie. Rośliny zainfekowane fitoplazmą wykazują szerokie spektrum objawów takich jak: wirescencja, fyllodia, żółknięcie, miotlastość zwijanie liści i zamieranie (Bertaccini i wsp. 2005) oraz karłowacenie, nieprawidłowości w budowie kwiatów i obniżoną żywotność, często prowadzącą do śmierci rośliny (Al-Zadjali i wsp. 2007).
Celem tego badania była identyfikacja i charakterystyka czynnika sprawczego choroby badanych pomidorów.
Materiały i metody
Próbki do badań
Próbki do badań zostały zebrane latem 2006 (Soly06gh) i 2007 (Soly07fi) z dwóch roślin pomidora zebranych odpowiednio, ze szklarni i z pola uprawnego w zachodniej Polsce. Badane próbki roślin były badane na obecność bakterii, grzybów i wirusów przy użyciu standardowych podłoży mikrobiologicznych, mikroskopu optycznego, elektronowego i testów biologicznych. Obecność fitoplazm w badanych próbkach badana była przy użyciu izolacji kwasów nukleinowych i wykrywaniu PCR ze starterami uniwersalnymi dla fitoplazm.
Izolacja DNA i warunki PCR
3,5g nerwów liściowych oraz całe, małe liście pomidora były pobrane z chorych roślin. Całkowity genomowy DNA został wyizolowany z badanych próbek roślin przy użyciu zmodyfikowanego protokołu CTAB (Padovan i wsp. 2000). Zewnętrzny PCR wykonano przy użyciu uniwersalnych dla fitoplazm starterów P1/P7, do amplifikacji rybosomalnego genu 16S rDNA. Produkt PCR (1.8 kb) rozcieńczono (1:30) sterylną wodą destylowaną i 1ml rozcieńczonego DNA użyto jako matrycę do wewnętrznego PCR, z wykorzystaniem pary starterów R16F2n/R16R2 (1,2kb) (Lee i wsp. 1998). Obie reakcje PCR wykonano przy użyciu polimerazy Taq DNA, w następujących warunkach: Zewnętrzny PCR ze starterami P1/P7, po wstępnej denaturacji w 94°C przez 2 min, przeprowadzono w 35 cyklach. Każdy cykl obejmował: denaturację prowadzoną w 94°C przez 30 sek., przyłączanie w 55°C przez 30 sek. i wydłużanie w 72°C przez 30 sek. Końcowe wydłużanie przeprowadzono w 72°C przez 7 min. Zagnieżdżony (ang. nested) PCR ze starterami R16F2n/R16R2, po wstępnej denaturacji w 94°C przez 2 min, został przeprowadzony w 35 cyklach, każdy w 94°C przez 1 min, 50°C przez 45 sek. i 72°C przez 1 min. Ostatni etap to wydłużanie w 72°C przez 10 min. Obie mieszaniny reakcyjne (25 ml) zawierały 20 ng genomowego DNA, 1 pmol każdego ze starterów, 1 jednostkę polimerazy Taq DNA i końcowe stężenie 0,2mM dNTP, 5mM MgCl2 oraz 1 x bufor do PCR. Jako pozytywnej i negatywnej kontroli reakcji użyto DNA wyizolowanego z Vinca sp. zainfekowanego fitoplazmą należącą do podgrupy 16SrI-C oraz DNA ze zdrowej rośliny pomidora.
Klonowanie produktów PCR
Namnożone fragmenty DNA (1,2 kb) fitoplazmatycznego rDNA zostały wklonowane do komórek Escherichia coli przy użyciu zestawu pGEM-T Easy Vector System II (Promega, Madison, WI, USA) według zaleceń producenta. Zrekombinowane plazmidy sprawdzono pod kątem obecności wklonowanego insertu przy użyciu płytek wskaźnikowych zawierających IPTG w pożywce. Po sprawdzeniu wszystkich zrekombinowanych komórek, te zawierające insert zostały przeniesione do 3 ml świeżej pożywki LB zawierajcego 3ml ampiciliny dodanej z roztworu o stężeniu 100 mg/ml. Po inkubacji w 37°C przez 18-24h z wytrząsaniem (160 rpm) namnożone komórki bakterii zostały wykorzystane do izolacji plazmidów przy użyciu zestawu QIAGEN MiniPrep Kit, według instrukcji producenta. Wyizolowane plazmidy badano na obecność insertu przy pomocy PCR ze starterami R16F2n/R16R2 oraz przez trawienie enzymem EcoRI. Po sprawdzeniu, plazmidy zawierające insert były sekwencjonowane przy użyciu uniwersalnych starterów M13.
Polimorfizm długości fragmentów restrykcyjnych (RFLP)
Analizę długości fragmentów restrykcyjnych końcowego produktu PCR o długości (1.2 kb) przeprowadzono przy użyciu następujących enzymów restrykcyjnych: MseI, AluI, HhaI, HpaII, RsaI i TaqI. Dla lepszego scharakteryzowania badanych sekwencji nukleotydowych przeprowadzono również wirtualną (in silico) analizę restrykcyjną przy użyciu programu ClustalW. Wyniki tej analizy przedstawiono w tabeli 2.
Analiza sekwencji
Uzyskane sekwencje nukleotydowe genu 16S rDNA badanych izolatów fitoplazm porównane były do innych grup fitoplazm dostępnych w GenBanku. Wyniki tego porównania przedstawiono w tabeli 3.
Sekwencjonowanie i analiza sekwencji
Sekwencje końcowych produktów obu reakcji PCR (1161bp i 1241bp) zostały zdeponowane w GenBanku i otrzymały numery akcesyjne (odpowiednio: EF164961 i EU402598). Ograniczona ilość sekwencji fitoplazmatycznych została pozyskana z GenBanku w celu badania podobieństwa i zależności filogenetycznych 2 Polskich izolatów i przedstawicieli fitoplazm z innych grup 16S rRNA. Wszystkie sekwencje pozyskane z GenBanku i wykorzystane w badaniach filogenetycznych wymieniono w tabeli 1. Przyrównanie sekwencji (ang. alignment) wykonano przy użyciu programu ClustalW. Wykonano analizy podobieństwa sekwencji i analizy filogenetyczne a ich wyniki przedstawione są w tabeli 3 oraz na rysunku 5. Dendrogram z analizą filogenetyczną wykonano przy użyciu metody maksymalnej parsymonii (ang. maximum parsimony). Analizy wykonano dla wartości bootstrap równej 1000 powtórzeń. Wszystkie analizy filogenetyczne wykonano przy użyciu programu Mega 3.1.
Tabela 1
Sekwencje genu 16S rDNA oraz międzygenowego odcinka 16S-23S rDNA fitoplazm wykorzystane w analizach filogenetycznych.
|
Numer akcesyjny
|
Oznaczenie |
Fitoplazma |
Grupa |
Grupa 16S rDNA |
Lit. |
|
EF164961 |
Soly06gh |
Tomato phyllody phytoplasma |
Aster yellows |
I-C |
- |
|
EU402598 |
Soly07fi |
Tomato phyllody phytoplasma |
Aster yellows |
I-C |
- |
|
AY863193 |
TBB-2 |
Tomato big bud phytoplasma |
Aster yellows |
I-A |
GB* |
|
AY863194 |
TBB-3 |
Tomato big bud phytoplasma |
Aster yellows |
I-A |
15 |
|
X68373 |
AAY |
American aster yellows phytoplasma |
Aster yellows |
I-B |
15 |
|
M86340 |
SAY |
Western aster yellows phytoplasma |
Aster yellows |
I-B |
15 |
|
AF222065 |
CPh-1 |
Clover phyllody phytoplasma |
Aster yellows |
I-C |
15, 4 |
|
AF222066 |
CPh-2 |
Clover phyllody phytoplasma |
Aster yellows |
I-C |
4 |
|
DQ851169 |
PWB-1 |
Paulownia witches'-broom phytoplasma |
Aster yellows |
I-D |
GB |
|
AY647460 |
PWB-2 |
Paulownia witches'-broom phytoplasma |
Aster yellows |
I |
GB |
|
AY265220 |
BBS-1 |
Blueberry stunt phytoplasma |
Aster yellows |
I-E1 |
GB |
|
AY265213 |
BBS-2 |
Blueberry stunt phytoplasma |
Aster yellows |
I-E2 |
GB |
|
L33765 |
PnWB |
Peanut witches'-broom mycoplasma-like organism |
Peanut WB |
II-A |
15 |
|
X83432 |
FBP |
Faba bean phyllody |
Peanut WB |
II-C |
15, 4 |
|
L04682 |
WX-1 |
Western X-disease mycoplasma-like organism |
X-disease |
III-A |
15 |
|
D12580 |
TWB |
Tsuwabuki witches'-broom |
X-disease |
III-B |
15 |
|
U18747 |
LY-4 |
Coconut lethal yellowing phytoplasma |
Coconut lethal yellows |
IV-A |
15 |
|
U18753 |
LDY |
Yucatan coconut lethal decline phytoplasma |
Coconut lethal yellows |
IV-B |
15 |
|
X80117 |
LDT |
Tanzanian coconut lethal decline phytoplasma |
Coconut lethal yellows |
IV-C |
15 |
|
X68376 |
ULW |
Elm witches`-broom |
Elm yellows |
V-A |
15 |
|
X76560 |
FD |
Flavescence doree |
Elm yellows |
V-C |
15, 4 |
|
AF228052 |
BLL |
Brinjal little leaf |
Clover proliferation |
VI |
4 |
|
X68339 |
AshY-2 |
Ash yellows phytoplasma |
Ash yellows |
VII-A |
15 |
|
AF353090 |
LofWB-1 |
Loofah witches'-broom phytoplasma |
Loofah WB |
VIII-A |
GB |
|
U18763 |
PPWB |
Caribbean PPWB mycoplasma-like organism |
Pigeon pea WB |
IX-A |
15 |
|
X68375 |
AP-3 |
Apple proliferation phytoplasma |
Apple proliferation |
X-A |
15 |
|
X76432 |
SCWL-2 |
Sugarcane white leaf phytoplasma |
Rice yellow dwarf |
XI-B |
15 |
|
X76429 |
BVK |
Leafhooper borne |
Rice yellow dwarf |
XI-C |
15 |
|
X76428 |
GY |
Grapevine yellows |
Stolbur |
XII-A |
15 |
|
L76865 |
AUSGY |
Australian grapevine yellows phytoplasma |
Stolbur |
XII-B |
15 |
|
AF248960 |
MPV |
Mexican periwinkle virescence phytoplasma |
MPV |
XIII |
2, 4 |
|
AF248961 |
BGWL-1 |
Bermuda grass white leaf phytoplasma |
Bermudagrass white leaf |
XIV |
2 |
|
AF147708 |
CaPBrz |
Hibiscus witches'-broom phytoplasma |
Ca. P. Brasiliense |
XV |
2, 4 |
|
DQ004910 |
- |
Acholeplasma palmae |
- |
Outgroup |
2 |
|
AY740427 |
- |
Acholeplasma palmae |
- |
Outgroup |
2 |
|
L33734 |
- |
Acholeplasma palmae |
- |
Outgroup |
2 |
* GB - odniesienie w bazie danych GenBanku.
Wyniki
Objawy chorobowe
Badane rośliny pomidora wykazywały objawy typowe dla infekcji fitoplazmatycznej takie jak: karłowacenie, skręcanie się pędów i liści oraz wirescencja (Rys. 1).
Rys. 1. Objawy chorobowe na pomidorze spowodowane przez izolat Soly06gh

Określenie czynnika sprawczego
Bakterie wyrosłe na pożywce TSA (ang. Tryptic Soy Agar) po bezpośredniej izolacji okazały się być epifitami i nie były patogenami roślin, co wykazano w przeprowadzonym teście nadwrażliwości na siewkach tytoniu i na roślinach pomidora. Obecność grzybów nie została wykryta na uniwersalnych pożywkach oraz pod mikroskopem optycznym. Testy biologiczne i w mikroskopie elektronowym nie wykazały obecności wirusów. W badanych roślinach pomidora wykazano jedynie obecność fitoplazm, poprzez uzyskanie prawidłowej wielkości produktu PCR i jego dalszą charakterystykę.
PCR
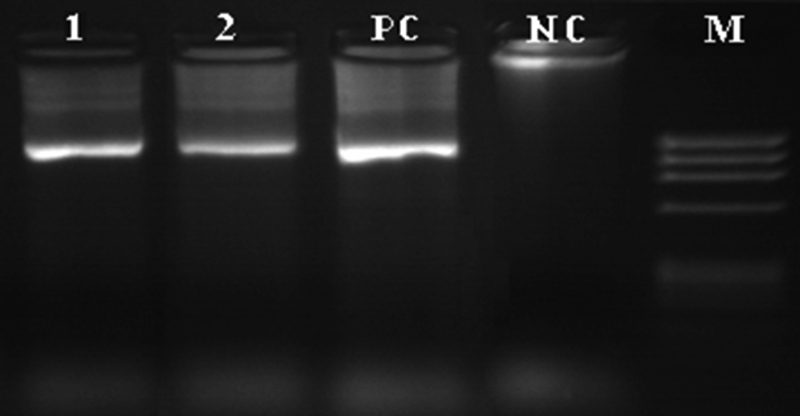
Produktem zewnętrznego PCR ze starterami P1/P7 był 1,8 kb fragment amplifikowanego genu 16S rDNA. W zagnieżdżonym PCR, ze starterami R16F2n/R16R2, uzyskano produkt o długości 1,2 kb należący do regionu międzygenowego 16S-23S rDNA w genomie fitoplazmy. Obecność prawidłowej wielkości produktów PCR wskazuje na to że obie badane rośliny zainfekowane były fitoplazmą (Rys. 2.).
Rys. 2. Obraz rozdziału elektroforycznego na żelu agarozowym (1,5%) obrazujący wielkości produktów PCR dla badanych fitoplazm. 1 - Izolat Soly06gh, 2 - Izoalt Soly07fi, PC - Kontrola pozytywna, NC - Kontrola negatywna, M - Marker DNA - ÆX174 DNA/BsuRI (HaeIII) Marker 9 (Fermentas)
RFLP
Profil restrykcyjny otrzymany produktu zagnieżdżonego PCR, po porównaniu z profilami kontrolnymi innych fitoplazm opublikowanych w literaturze (Rys. 4) oraz z profilami innych fitoplazm innych grup 16S rRNA (Lee i wsp. 1998) wskazywał że badane próbki roślin należą do grupy 16Sr I-C rRNA (Rys. 3 i rys. 4). Wirtualna analiza restrykcyjna wykazała, że izolat Soly06gh ma jedno miejsce restrykcyjne dla 2 enzymów: AseI i EcoRV i dlatego może on być odróżniony od innych fitoplazm w tej grupie (Tab. 2).
Tabela 2
Wyniki wirtualnej analizy restrykcyjnej sekwencji nukleotydowych produktów PCR uzyskanych z badanych izolatów fitoplazm oraz z wybranych szczepów fitoplazm należących do podgrupy 16S I-C rRNA i dostępnych w GenBanku
|
Enzym
|
Ilość miejsc restrykcyjnych |
|||||||
|
EF164961 |
EU402598 |
AF222065 |
AF222066 |
AY265217 |
AY265218 |
AF200431 |
AY953423 |
|
|
AluI |
5 |
5 |
5 |
4 |
4 |
4 |
5 |
4 |
|
HhaI |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
|
HpaII |
4 |
3 |
3 |
3 |
3 |
3 |
3 |
3 |
|
MseI |
9 |
8 |
8 |
9 |
9 |
9 |
8 |
9 |
|
RsaI |
7 |
7 |
7 |
7 |
7 |
7 |
7 |
7 |
|
TaqI |
2 |
2 |
2 |
2 |
2 |
2 |
2 |
2 |
|
AseI |
1 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
|
EcoRV |
1 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
|
BsiWI |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
|
Csp45I |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
|
Eco105I |
2 |
2 |
2 |
2 |
2 |
2 |
2 |
2 |
|
EcoRI |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
|
Hin1I |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
|
HincII |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
|
HpaI |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
|
KpnI |
2 |
2 |
2 |
2 |
2 |
2 |
2 |
2 |
|
NciI |
2 |
2 |
2 |
2 |
2 |
2 |
2 |
2 |
|
NspV |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
|
SacII |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
Rys. 3. Profil restrykcyjny izolatu kontrolnego i badanego izolatu Soly07fi. Od lewej: ÆX174 DNA Marker, Kontrola pozytywna - profil restrykcyjny podgrupy 16Sr I-C rRNA, enzymy: MseI, AluI, HhaI, HpaII, RsaI, TaqI, ÆX174 DNA Marker, Próbka Soly07fi - analogicznie
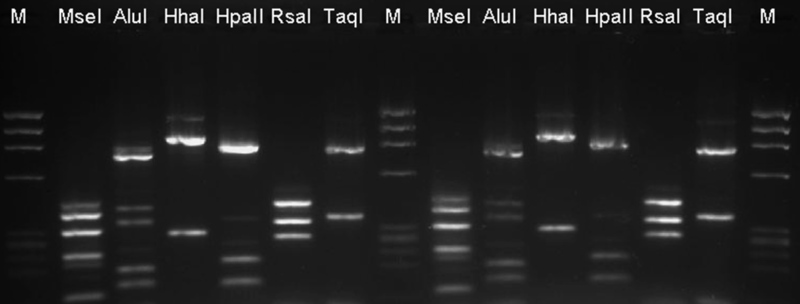
Rys. 4. Profil restrykcyjny izolatu Soly06gh. Od lewej: MseI, AluI, HhaI, HpaII, RsaI, TaqI, ÆX174 marker DNA
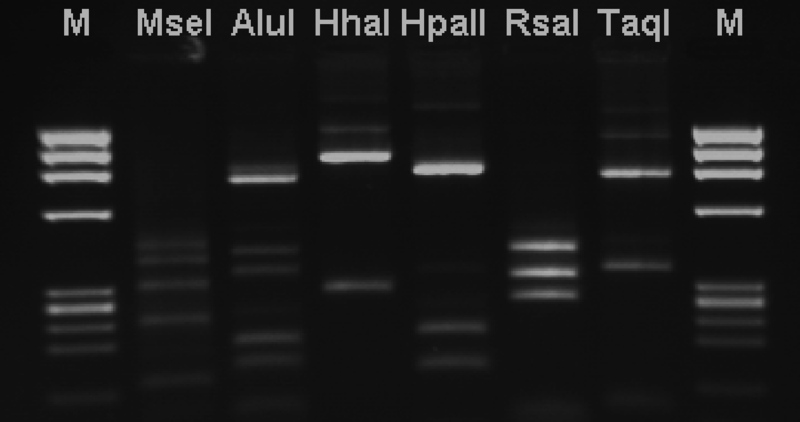
Analiza sekwencji
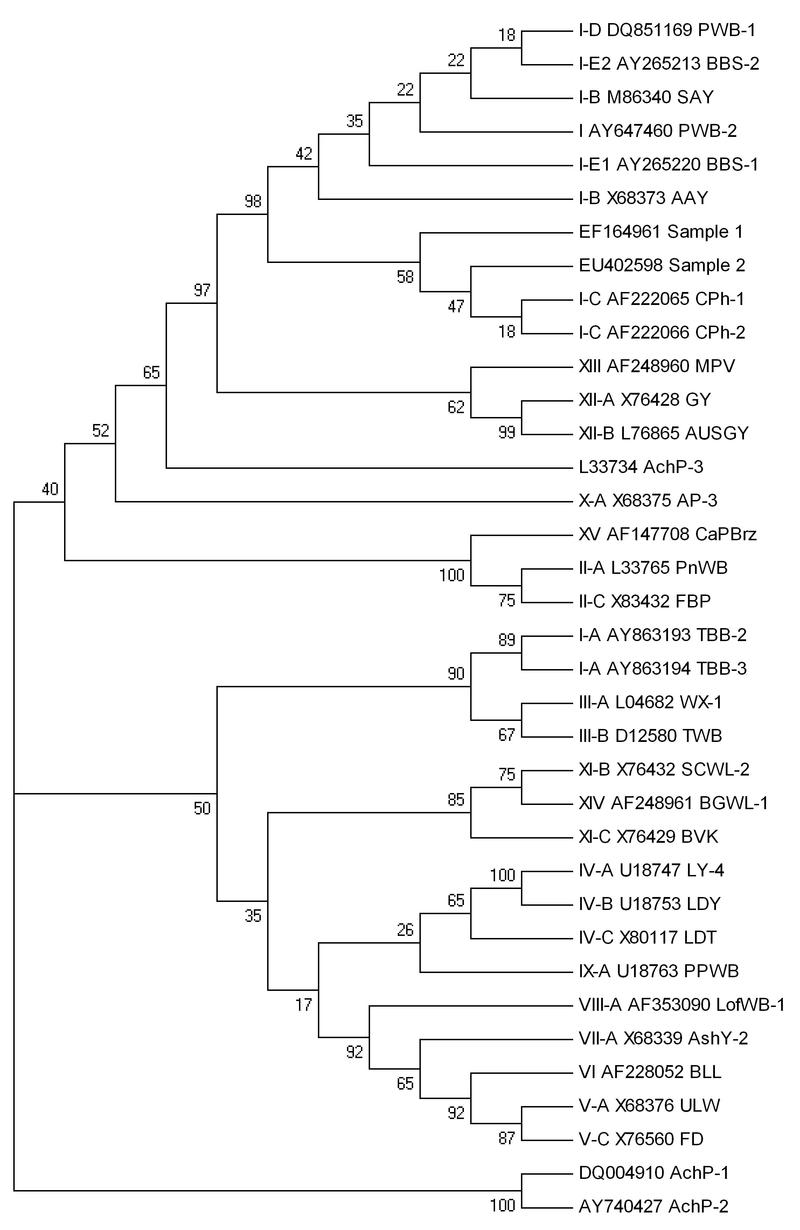
Wykonana analiza sekwencji genu 16S rDNA badanych izolatów i fitoplazm z innych grup (Tab. 3) potwierdziła, że badane izolaty należą do grupy 16S rRNA I-C. Homologia badanych sekwencji w obrębie podgrupy 16S rRNA I-C wynosiła 99% (Tab. 3). Wynik ten w całości potwierdzony został przez przeprowadzone analizy filogenetyczne (Rys. 5).
Tabela 3
Analiza podobieństwa sekwencji 16S rDNA pomiędzy badanymi izolatami i prezdstawicielami fitoplazm z innych grup.
|
Fitoplazma * |
Grupa |
|
1 |
2 |
3 |
4 |
5 |
6 |
7 |
8 |
9 |
10 |
11 |
12 |
13 |
14 |
15 |
16 |
17 |
18 |
19 |
20 |
21 |
22 |
23 |
24 |
25 |
26 |
27 |
28 |
29 |
30 |
31 |
32 |
33 |
|
Soly06gh |
I-C |
1 |
ID |
0,990 |
0,895 |
0,896 |
0,984 |
0,987 |
0,990 |
0,990 |
0,987 |
0,987 |
0,988 |
0,987 |
0,894 |
0,894 |
0,897 |
0,900 |
0,901 |
0,896 |
0,894 |
0,894 |
0,888 |
0,894 |
0,894 |
0,900 |
0,886 |
0,917 |
0,903 |
0,906 |
0,953 |
0,956 |
0,957 |
0,899 |
0,891 |
|
Soly07fi |
I-C |
2 |
0,990 |
ID |
0,898 |
0,899 |
0,987 |
0,991 |
0,998 |
0,998 |
0,990 |
0,991 |
0,993 |
0,992 |
0,897 |
0,899 |
0,901 |
0,903 |
0,904 |
0,900 |
0,897 |
0,898 |
0,892 |
0,898 |
0,896 |
0,905 |
0,891 |
0,923 |
0,906 |
0,909 |
0,960 |
0,962 |
0,961 |
0,902 |
0,896 |
|
TBB-2 |
I-A |
3 |
0,895 |
0,898 |
ID |
0,995 |
0,891 |
0,895 |
0,898 |
0,900 |
0,892 |
0,893 |
0,895 |
0,895 |
0,907 |
0,901 |
0,987 |
0,992 |
0,933 |
0,929 |
0,937 |
0,925 |
0,920 |
0,930 |
0,921 |
0,926 |
0,930 |
0,898 |
0,943 |
0,940 |
0,885 |
0,891 |
0,896 |
0,937 |
0,904 |
|
TBB-3 |
I-A |
4 |
0,896 |
0,899 |
0,995 |
ID |
0,892 |
0,896 |
0,899 |
0,901 |
0,893 |
0,894 |
0,896 |
0,896 |
0,906 |
0,900 |
0,986 |
0,991 |
0,932 |
0,928 |
0,936 |
0,924 |
0,919 |
0,929 |
0,921 |
0,925 |
0,929 |
0,897 |
0,942 |
0,939 |
0,886 |
0,890 |
0,896 |
0,936 |
0,904 |
|
AAY |
I-B |
5 |
0,984 |
0,987 |
0,891 |
0,892 |
ID |
0,994 |
0,987 |
0,987 |
0,990 |
0,991 |
0,991 |
0,990 |
0,891 |
0,893 |
0,894 |
0,897 |
0,897 |
0,892 |
0,892 |
0,891 |
0,885 |
0,891 |
0,892 |
0,898 |
0,882 |
0,919 |
0,899 |
0,902 |
0,955 |
0,956 |
0,957 |
0,895 |
0,889 |
|
SAY |
I-B |
6 |
0,987 |
0,991 |
0,895 |
0,896 |
0,994 |
ID |
0,991 |
0,991 |
0,993 |
0,994 |
0,994 |
0,993 |
0,893 |
0,895 |
0,897 |
0,900 |
0,899 |
0,895 |
0,895 |
0,893 |
0,887 |
0,893 |
0,895 |
0,901 |
0,885 |
0,920 |
0,902 |
0,904 |
0,958 |
0,959 |
0,961 |
0,897 |
0,892 |
|
CPh-1 |
I-C |
7 |
0,990 |
0,998 |
0,898 |
0,899 |
0,987 |
0,991 |
ID |
0,998 |
0,990 |
0,991 |
0,993 |
0,992 |
0,897 |
0,899 |
0,901 |
0,903 |
0,903 |
0,899 |
0,899 |
0,899 |
0,894 |
0,898 |
0,896 |
0,907 |
0,892 |
0,925 |
0,906 |
0,909 |
0,960 |
0,962 |
0,961 |
0,902 |
0,897 |
|
CPh-2 |
I-C |
8 |
0,990 |
0,998 |
0,900 |
0,901 |
0,987 |
0,991 |
0,998 |
ID |
0,990 |
0,991 |
0,993 |
0,992 |
0,899 |
0,901 |
0,903 |
0,905 |
0,905 |
0,901 |
0,899 |
0,899 |
0,894 |
0,899 |
0,898 |
0,907 |
0,892 |
0,925 |
0,908 |
0,910 |
0,960 |
0,962 |
0,962 |
0,903 |
0,896 |
|
PWB-1 |
I-D |
9 |
0,987 |
0,990 |
0,892 |
0,893 |
0,990 |
0,993 |
0,990 |
0,990 |
ID |
0,995 |
0,993 |
0,993 |
0,892 |
0,894 |
0,895 |
0,897 |
0,897 |
0,893 |
0,893 |
0,892 |
0,886 |
0,892 |
0,893 |
0,899 |
0,883 |
0,919 |
0,900 |
0,903 |
0,957 |
0,958 |
0,960 |
0,896 |
0,890 |
|
PWB-2 |
I |
10 |
0,987 |
0,991 |
0,893 |
0,894 |
0,991 |
0,994 |
0,991 |
0,991 |
0,995 |
ID |
0,994 |
0,993 |
0,895 |
0,896 |
0,896 |
0,898 |
0,899 |
0,895 |
0,895 |
0,892 |
0,886 |
0,892 |
0,894 |
0,900 |
0,884 |
0,920 |
0,901 |
0,903 |
0,958 |
0,959 |
0,961 |
0,896 |
0,892 |
|
BBS-1 |
I-E1 |
11 |
0,988 |
0,993 |
0,895 |
0,896 |
0,991 |
0,994 |
0,993 |
0,993 |
0,993 |
0,994 |
ID |
0,997 |
0,896 |
0,898 |
0,897 |
0,900 |
0,900 |
0,897 |
0,896 |
0,894 |
0,888 |
0,894 |
0,894 |
0,902 |
0,887 |
0,920 |
0,903 |
0,905 |
0,958 |
0,959 |
0,961 |
0,898 |
0,894 |
|
BBS-2 |
I-E2 |
12 |
0,987 |
0,992 |
0,895 |
0,896 |
0,990 |
0,993 |
0,992 |
0,992 |
0,993 |
0,993 |
0,997 |
ID |
0,895 |
0,896 |
0,897 |
0,900 |
0,900 |
0,896 |
0,896 |
0,893 |
0,887 |
0,893 |
0,894 |
0,901 |
0,885 |
0,920 |
0,903 |
0,905 |
0,958 |
0,959 |
0,961 |
0,898 |
0,892 |
|
PnWB |
II-A |
13 |
0,894 |
0,897 |
0,907 |
0,906 |
0,891 |
0,893 |
0,897 |
0,899 |
0,892 |
0,895 |
0,896 |
0,895 |
ID |
0,979 |
0,909 |
0,911 |
0,905 |
0,905 |
0,904 |
0,887 |
0,883 |
0,896 |
0,898 |
0,903 |
0,899 |
0,890 |
0,906 |
0,905 |
0,888 |
0,890 |
0,893 |
0,902 |
0,966 |
|
FBP |
II-C |
14 |
0,894 |
0,899 |
0,901 |
0,900 |
0,893 |
0,895 |
0,899 |
0,901 |
0,894 |
0,896 |
0,898 |
0,896 |
0,979 |
ID |
0,903 |
0,906 |
0,899 |
0,898 |
0,898 |
0,881 |
0,877 |
0,889 |
0,891 |
0,898 |
0,897 |
0,889 |
0,899 |
0,901 |
0,892 |
0,895 |
0,895 |
0,896 |
0,965 |
|
WX-1 |
III-A |
15 |
0,897 |
0,901 |
0,987 |
0,986 |
0,894 |
0,897 |
0,901 |
0,903 |
0,895 |
0,896 |
0,897 |
0,897 |
0,909 |
0,903 |
ID |
0,990 |
0,933 |
0,930 |
0,939 |
0,928 |
0,922 |
0,931 |
0,924 |
0,930 |
0,931 |
0,901 |
0,943 |
0,943 |
0,886 |
0,894 |
0,896 |
0,938 |
0,906 |
|
TWB |
III-B |
16 |
0,900 |
0,903 |
0,992 |
0,991 |
0,897 |
0,900 |
0,903 |
0,905 |
0,897 |
0,898 |
0,900 |
0,900 |
0,911 |
0,906 |
0,990 |
ID |
0,938 |
0,934 |
0,942 |
0,928 |
0,923 |
0,933 |
0,925 |
0,933 |
0,932 |
0,904 |
0,947 |
0,944 |
0,892 |
0,896 |
0,901 |
0,943 |
0,909 |
|
LY-4 |
IV-A |
17 |
0,901 |
0,904 |
0,933 |
0,932 |
0,897 |
0,899 |
0,903 |
0,905 |
0,897 |
0,899 |
0,900 |
0,900 |
0,905 |
0,899 |
0,933 |
0,938 |
ID |
0,992 |
0,967 |
0,950 |
0,946 |
0,951 |
0,943 |
0,946 |
0,939 |
0,908 |
0,957 |
0,953 |
0,892 |
0,894 |
0,903 |
0,951 |
0,897 |
|
LDY |
IV-B |
18 |
0,896 |
0,900 |
0,929 |
0,928 |
0,892 |
0,895 |
0,899 |
0,901 |
0,893 |
0,895 |
0,897 |
0,896 |
0,905 |
0,898 |
0,930 |
0,934 |
0,992 |
ID |
0,965 |
0,949 |
0,945 |
0,949 |
0,940 |
0,944 |
0,937 |
0,906 |
0,952 |
0,950 |
0,890 |
0,891 |
0,901 |
0,947 |
0,896 |
|
LDT |
IV-C |
19 |
0,894 |
0,897 |
0,937 |
0,936 |
0,892 |
0,895 |
0,899 |
0,899 |
0,893 |
0,895 |
0,896 |
0,896 |
0,904 |
0,898 |
0,939 |
0,942 |
0,967 |
0,965 |
ID |
0,951 |
0,946 |
0,950 |
0,941 |
0,945 |
0,945 |
0,908 |
0,956 |
0,955 |
0,885 |
0,889 |
0,895 |
0,954 |
0,899 |
|
ULW |
V-A |
20 |
0,894 |
0,898 |
0,925 |
0,924 |
0,891 |
0,893 |
0,899 |
0,899 |
0,892 |
0,892 |
0,894 |
0,893 |
0,887 |
0,881 |
0,928 |
0,928 |
0,950 |
0,949 |
0,951 |
ID |
0,989 |
0,967 |
0,957 |
0,955 |
0,933 |
0,898 |
0,948 |
0,945 |
0,879 |
0,886 |
0,887 |
0,945 |
0,888 |
|
FD |
V-C |
21 |
0,888 |
0,892 |
0,920 |
0,919 |
0,885 |
0,887 |
0,894 |
0,894 |
0,886 |
0,886 |
0,888 |
0,887 |
0,883 |
0,877 |
0,922 |
0,923 |
0,946 |
0,945 |
0,946 |
0,989 |
ID |
0,963 |
0,953 |
0,952 |
0,930 |
0,897 |
0,945 |
0,941 |
0,874 |
0,880 |
0,883 |
0,942 |
0,884 |
|
BLL |
VI |
22 |
0,894 |
0,898 |
0,930 |
0,929 |
0,891 |
0,893 |
0,898 |
0,899 |
0,892 |
0,892 |
0,894 |
0,893 |
0,896 |
0,889 |
0,931 |
0,933 |
0,951 |
0,949 |
0,950 |
0,967 |
0,963 |
ID |
0,969 |
0,962 |
0,935 |
0,899 |
0,953 |
0,948 |
0,883 |
0,887 |
0,896 |
0,951 |
0,890 |
|
AshY-2 |
VII-A |
23 |
0,894 |
0,896 |
0,921 |
0,921 |
0,892 |
0,895 |
0,896 |
0,898 |
0,893 |
0,894 |
0,894 |
0,894 |
0,898 |
0,891 |
0,924 |
0,925 |
0,943 |
0,940 |
0,941 |
0,957 |
0,953 |
0,969 |
ID |
0,957 |
0,935 |
0,901 |
0,951 |
0,945 |
0,888 |
0,889 |
0,892 |
0,949 |
0,893 |
|
LofWB-1 |
VIII-A |
24 |
0,900 |
0,905 |
0,926 |
0,925 |
0,898 |
0,901 |
0,907 |
0,907 |
0,899 |
0,900 |
0,902 |
0,901 |
0,903 |
0,898 |
0,930 |
0,933 |
0,946 |
0,944 |
0,945 |
0,955 |
0,952 |
0,962 |
0,957 |
ID |
0,934 |
0,912 |
0,945 |
0,942 |
0,893 |
0,897 |
0,897 |
0,948 |
0,898 |
|
PPWB |
IX-A |
25 |
0,886 |
0,891 |
0,930 |
0,929 |
0,882 |
0,885 |
0,892 |
0,892 |
0,883 |
0,884 |
0,887 |
0,885 |
0,899 |
0,897 |
0,931 |
0,932 |
0,939 |
0,937 |
0,945 |
0,933 |
0,930 |
0,935 |
0,935 |
0,934 |
ID |
0,901 |
0,941 |
0,939 |
0,885 |
0,886 |
0,891 |
0,945 |
0,894 |
|
AP-3 |
X-A |
26 |
0,917 |
0,923 |
0,898 |
0,897 |
0,919 |
0,920 |
0,925 |
0,925 |
0,919 |
0,920 |
0,920 |
0,920 |
0,890 |
0,889 |
0,901 |
0,904 |
0,908 |
0,906 |
0,908 |
0,898 |
0,897 |
0,899 |
0,901 |
0,912 |
0,901 |
ID |
0,911 |
0,910 |
0,918 |
0,921 |
0,924 |
0,912 |
0,893 |
|
SCWL-2 |
XI-B |
27 |
0,903 |
0,906 |
0,943 |
0,942 |
0,899 |
0,902 |
0,906 |
0,908 |
0,900 |
0,901 |
0,903 |
0,903 |
0,906 |
0,899 |
0,943 |
0,947 |
0,957 |
0,952 |
0,956 |
0,948 |
0,945 |
0,953 |
0,951 |
0,945 |
0,941 |
0,911 |
ID |
0,978 |
0,891 |
0,896 |
0,905 |
0,982 |
0,898 |
|
BVK |
XI-C |
28 |
0,906 |
0,909 |
0,940 |
0,939 |
0,902 |
0,904 |
0,909 |
0,910 |
0,903 |
0,903 |
0,905 |
0,905 |
0,905 |
0,901 |
0,943 |
0,944 |
0,953 |
0,950 |
0,955 |
0,945 |
0,941 |
0,948 |
0,945 |
0,942 |
0,939 |
0,910 |
0,978 |
ID |
0,893 |
0,895 |
0,903 |
0,976 |
0,898 |
|
GY |
XII-A |
29 |
0,953 |
0,960 |
0,885 |
0,886 |
0,955 |
0,958 |
0,960 |
0,960 |
0,957 |
0,958 |
0,958 |
0,958 |
0,888 |
0,892 |
0,886 |
0,892 |
0,892 |
0,890 |
0,885 |
0,879 |
0,874 |
0,883 |
0,888 |
0,893 |
0,885 |
0,918 |
0,891 |
0,893 |
ID |
0,978 |
0,961 |
0,890 |
0,887 |
|
AUSGY |
XII-B |
30 |
0,956 |
0,962 |
0,891 |
0,890 |
0,956 |
0,959 |
0,962 |
0,962 |
0,958 |
0,959 |
0,959 |
0,959 |
0,890 |
0,895 |
0,894 |
0,896 |
0,894 |
0,891 |
0,889 |
0,886 |
0,880 |
0,887 |
0,889 |
0,897 |
0,886 |
0,921 |
0,896 |
0,895 |
0,978 |
ID |
0,962 |
0,894 |
0,890 |
|
MPV |
XIII |
31 |
0,957 |
0,961 |
0,896 |
0,896 |
0,957 |
0,961 |
0,961 |
0,962 |
0,960 |
0,961 |
0,961 |
0,961 |
0,893 |
0,895 |
0,896 |
0,901 |
0,903 |
0,901 |
0,895 |
0,887 |
0,883 |
0,896 |
0,892 |
0,897 |
0,891 |
0,924 |
0,905 |
0,903 |
0,961 |
0,962 |
ID |
0,903 |
0,889 |
|
BGWL-1 |
XIV |
32 |
0,899 |
0,902 |
0,937 |
0,936 |
0,895 |
0,897 |
0,902 |
0,903 |
0,896 |
0,896 |
0,898 |
0,898 |
0,902 |
0,896 |
0,938 |
0,943 |
0,951 |
0,947 |
0,954 |
0,945 |
0,942 |
0,951 |
0,949 |
0,948 |
0,945 |
0,912 |
0,982 |
0,976 |
0,890 |
0,894 |
0,903 |
ID |
0,893 |
|
CaPBrz |
XV |
33 |
0,891 |
0,896 |
0,904 |
0,904 |
0,889 |
0,892 |
0,897 |
0,896 |
0,890 |
0,892 |
0,894 |
0,892 |
0,966 |
0,965 |
0,906 |
0,909 |
0,897 |
0,896 |
0,899 |
0,888 |
0,884 |
0,890 |
0,893 |
0,898 |
0,894 |
0,893 |
0,898 |
0,898 |
0,887 |
0,890 |
0,889 |
0,893 |
ID |
* Skróty nazw fitoplazm wyjaśniono w tabeli 1.
Rys. 5. Dendrogram przedstawiający wyniki analizy filogenetycznej fragmentów genu 16S rRNA skostruowany przy użyciu metody neighbor joining. Polskie izolaty oznaczono czarnym kwadratem. Pozostałe sekwencje pochodzą z bazy danych GenBank. Sekwencje analizowano przy użyciu programów: BioEdit i Mega 3.1. Przyrównanie sekwencji wykonano przy użyciu programu ClustalW. Dla powyższej analizy przyjęto wartość bootstrap równą 1000.
Dyskusja
Choroby fitoplazmatyczne na pomidorze były wcześniej opisywane w wielu krajach, takich jak: Boliwia (Arocha i wsp. 2007), Brazylia (Mello i wsp. 2006), Egipt (El-banna i wsp. 2007) Francja (Pracros i wsp. 2006), Grecja (Vellios i Lioliopoulou 2007), Włochy (Del Serrone i wsp. 2001), Jordania (Ghandi i wsp. 2003), Liban (Coueiri i wsp. 2007), Turcja (Ozdemir i wsp. 2009) i Stany Zjednoczone (Dale i Smith 1975). Jako czynnik sprawczy wymieniane były grupy: I, III, V, VI i XII, 16S rRNA (Del Serrone i wsp. 2001). Spośród nich grupa 16S-I rRNA (Candidatus Phytoplasma asteris (Lee i wsp. 2004) jest najczęstszą przyczyną infekcji roślin pomidora. W obrębie tej grupy, w roli patogenu pomidora wymieniano dotychczas tylko Tomato Big Bud Phytoplasma (TBB) należącą do podgrupy 16S rRNA I-A (Dale i Smith 1975). Oba badane profile fitoplazm wykazywały ten sam wzór/profil restrykcyjny, charakterystyczny dla podgrupy 16S rRNA I-C (Lee i wsp. 1998). Wirtualna analiza restrykcyjna wykazała minimalne różnice w sekwencji obu badanych szczepów, które mogą być wykorzystane do odróżnienia ich od siebie (Tab. 2). Niniejsza praca opisuje dwa pierwsze doniesienia o wykryciu i zidentyfikowaniu fitoplazmy z podgrupy 16S rRNA I-C jako sprawcy choroby pomidora. Jest to fakt nowy dla nauki i opisany po raz pierwszy nie tylko w Polsce, ale i na świecie.
Zakres gospodarzy dla fitoplazm z podgrupy 16S rRNA I-C jest szeroki. Dotychczas wykazano, że oprócz pomidora infekują one również następujące rośliny: koniczyna (Bertaccini i wsp. 2005), lilie (Poncarova-Vorackova i wsp. 1998), cyklameny (Alma i wsp. 2000), grusze i jabłonie (Franova i wsp. 2008), Limonium sinatum (Babaie i wsp. 2007), winogrona (Riolo i wsp. 2006) i truskawki (Franova i Petrzik 2008).
Literatura
- Alma A, Marzachi C, d`Aquillo M, Bosco D. (2000) Cyklamen (Cyklamen persicum L.): a dead end host species for 16Sr-IB and –IC subgroup phytoplasmas. Ann Appl Biol 136:173-178
- Al-Zadjali AD, Natsuaki T, Okuda S. (2007) Detection, Identification and Molecular Characterization of Phytolasma Associated with Arabian jasmine (Jasminum sambac L.) Witches`Broom in Oman. J Phytopathol 155:211-219
- Arocha Y, Antesana O, Montellano E, Franco P, Plata G, Jones P. (2007) ‘Candidatus Phytoplasma lycopersici’, a phytoplasma associated with ‘hoja de perejil’ disease in Bolivia Int J Syst Evol Microbiol 57:1704-1710
- Arocha Y, Lopez M, Fernandez M, Pinol B, Horta D, Peralta EL, Almeida R Caravajal O, Picornell S, Wilson MR, Jones P. (2005) Transmission of a sugarcane yellow leaf phytoplasma by the delaphacid planthooper Saccharosydne saccharivora, a new vector of sugarcane yellow leaf syndrome. Plant Dis 54:634-642
- Babaie G, Khatabi B, Bayat H, Rastgou M, Hosseini A, Salekdeh GH. (2007) Detection and Characterization of Phytoplasmas Infecting Ornamental and Weed Plants in Iran. J Phytopathol 155:368-372
- Bertaccini A, Franova J, Botti S, Tabanelli D, (2005) Molecular characterization of phytoplasmas in lilies with fasciation in the Czech Republic. FEMS Microbiol Lett 249:79-85
- Chiykowski LN. (1991) Vector-pathogenhost plant relationships of clover phyllody mycoplasmalike organism and the vector leafhopper Paraphlepsius irroratus. Can. J. Plant Pathol. 13:11-18
- Chiykowski LN, Sinha RC. (1989) Differentiation of MLO disease by means of symptomatology and vector transmission. Zentralbl. Bakteriol. Hyg. Suppl. 20:280-287
- Coueiri E, Salar P, Jreijiri F, El Zamar S, Massad R, Abdul-Nour H, Bove JM, Danet JL, Foissac X. (2007) Occurence and distribution of “Candidatus Phytoplasma trifolii” associated with diseases of solanacearus crops in Lebanon. Eur J Plant Pathol 118:411-416
- Dale JL, Smith LD. (1975) Mycoplasma-like bodies observed in tomato plants with big bud in Arkansas. Plant Dis Rept 59:455-458
- Del Serrone P, Marzachi C, Brangaloni M, Galeffi P. (2001) Phytoplasma infection of tomato in central Italy. Phytopathol Mediterr 40:137-142
- El-banna OH, Mikhail MS, Farag AG, Mohammed AMS. (2007) Detection of phytoplasma in tomato and pepper plants by electron microscopy and molecular biology based methods. Egyptian J Virology 4:93-111
- Franova J, Petrzik K. (2008) Strawberry sterility – what is the causal agent(s)? Acta Hort (ISHS) 780:27-34
- Franova J, Petrzik K, Paprstein F, Kucerova J, Ruzickova M. (2008) Molecular identification of phytoplasmas in cultivar collection and production plantations of apple and pear trees in the Czech Republic. Acta Hort (ISHS) 781:359-368
- Ghandi HA, Amjad BK, Isam F. (2003) Detection and Molecular Characterization of a Phytoplasma Associated with Big Bud Disease of Tomatoes in Jordan. J Phytopathol 151:223–227
- Lee IM, Gundersen-Rindal DE, Davis RE, Bartoszyk IM. (1998) Revised classification scheme of phytoplasmas based on RFLP analyses of 16S rRNA and ribosomal protein gene sequences. Int J Syst Bacteriol 48:1153-1169
- Lee IM, Davis RE, Gundersen-Rindal DE. (2000) Phytoplasma: phytopathogenic mollicutes. Annu Rev Microbiol 54:221–255
- Lee IM, Davis RE, Fletcher J. (2001) Cell-wall free bacteria. In: Laboratory Guide for Identification of Plant Pathogenic Bacteria. Schaad NW, Jones JB, Chun W (eds.) St. Paul, Minnesota, USA, The American Phytopathology Society Press, pp. 283 – 320
- Lee IM, Gundersen-Rindal DE, Davis RE, Bottner KD, Marcone C, Seemuller E. (2004) ‘Candidatus Phytoplasma asteris’, a novel phytoplasma taxon associated with aster yellows and related diseases. Int J Syst Evol Micr 54:1037–1048
- McCoy RE, Caudwell A, Chang CJ. (1989). Plant diseases associated with mycoplasma-like organisms. In: The Mycoplasmas. Whitcomb RF, Tully JG, (eds.) San Diego CA, USA, Academic Press pp. 545–640
- Mello A, Bedendo IP, Camargo LE. (2006) Sequence heterogeneity in the 16S rDNA of tomato big bud phytoplasma belonging to group 16SrIII. J Phytopathol 154:245-249
- Ozdemir N, Saygili H, Sahin F, Karsavuran Y, Bayrak OF, Oral B. (2009) Host range and genetic characterization of a phytoplasma causing tomato stolbur disease in Turkey. Acta Hort (ISHS) 808:255-262
- Padovan AC, Firrao G, Schneider B, Gibb KS. (2000) Chromosome mapping of the sweet potato little leaf phytoplasma reveals genome heterogeneity within the phytoplasmas. Microbiol 146:893-902
- Palmano S. (2001) A comparison of different phytoplasma DNA extraction methods using competitive PCR. Phytopathol Mediterr 40:99-107
- Poncarova-Vorackova Z, Franova J, Valova P, Mertelik J, Navratil J, Nebesarova J. (1998) Identification of Phytoplasma infecting Lilium mantragon in the Czech Republic. J Phytopathol 146:609-612
- Pospieszny H, Krawczyk K, Kamasa J, Petrzik K. (2007) First Report of a Phytoplasma Affecting Tomato in Poland. Plant Dis. 91:1054
- Pracros P, Renaudin J, Eveillard S, Mouras A, Hernould M. (2006) Tomato Flower Abnormalities Induced by Stolbur Phytoplasma Infection Are Associated with Changes of Expression of Floral Development Genes. Mol Plant Microbe Interact 19:62-68
- Riolo P, Murolo S, Riga F, Nardi S, Isidoro N, Romanazzi G. (2006) Identification of phytoplasmas in vectors and potential vectors of grapevine yellows in Marche region, Central-Eastern Italy. Giornate Fitopatologiche 2:27-29
- Shiomi T, Sugiura M. (1984) Grouping of mycoplasma-like organisms transmitted by the leafhopper vector, Macrosteles orientalis Virvaste, based on host range. Ann. Phytopathol. Soc. Jpn 50:149-157
- Vellios E, Liolipoulou F. (2007) Detection and characterization of phytoplasmas infecting tomato plants in Grece. Bull Insectol 60:157-158