Xanthomonas campestris pv. holcicola, Xanthomonas campestris pv. zeae - bakteryjna plamistość liści (ang. bacterial leaf spot).
Xanthomonas campestris pv. holcicola (Elliott) Dye, Xanthomonas campestris pv. zeae - bakteryjna plamistość liści (ang. bacterial leaf spot).
Występowanie i zakres gospodarzy
Występowanie tych gatunków bakterii jest ograniczone do południa Afryki (RPA) (Giester i wsp. 2004, Claflin 2000, Coutinho i Wallis 1991). Dla X. campestris pv. zeae, kukurydza jest jedynym znanym żywicielem, natomiast sam X. campestris jest polifagiem i powoduje choroby wielu gatunków roślin (Claflin 2000).
Rozprzestrzenianie i szkodliwość
Bakteryjna plamistość liści kukurydzy spowodowana przez X. campestris pv zeae jest często opisywana w literaturze naukowej (Giester i wsp. 2004, Claflin 2000, Coutinho i Wallis 1991). Bakteria ta do wniknięcia w roślinę nie wymaga ran na jej powierzchni, gdyż zwykle wnika przez aparaty szparkowe i następnie przemieszcza się w przestrzeniach międzykomórkowych do komórek ksylemu. Namnażające się masy bakterii mogą zablokować jego elementy, co prowadzi do zahamowania transportu wody w roślinie i więdnięcia roślin. Jednakże więdnięcie całych roślin w warunkach polowych jest rzadko obserwowane. Chwasty sąsiadujące z polami kukurydzy mogą być dla tej bakterii gospodarzami alternatywnymi.
Objawy charakterystyczne dla X. campestris to tworzące się na liściach żółtobrązowe martwice, szerokie na 2-3 mm z falistymi i nieregularnymi żółtymi brzegami, biegnące równolegle do nerwów liściowych. Plamy mogą się rozciągać na całą długość liści i często zlewają się by formować duże nekrotyczne obszary, które bardzo przypominają objawy suszy. Więdnięcie roślin jest rzadko obserwowane. Żółte kropelki wydzieliny bakteryjnej mogą się formować na plamach w warunkach wysokiej wilgotności.
Rola pospolicie występujących chwastów polnych, jako potencjalnego rezerwuaru bakterii fitopatogenicznych opisana została w literaturze również w odniesieniu do gatunku X. campestris, patogena kukurydzy oraz sprawcy chorób roślin krzyżowych (Cruciferaceae). Wykazano, że częstość występowania tego gatunku patogena w chwastach jest wyższa na terenach bezpośrednio przylegających do pól hodowlanych, na których uprawiane są warzywa w celach komercyjnych (Ignatov i wsp. 2007, Schaad i Diagnese 1981). W świetle tych wyników szczególnego znaczenia w ochronie upraw przed chorobami bakteryjnymi nabiera kontrola stanu zachwaszczenia pól.
Wykrywanie i identyfikacja
Rodzaj Xanthomonas to pojedyncze komórki, proste pałeczki o wymiarach od 0,2 - 0,8 µm do 0,6-2,0 µm zwykle 0,4-1,0 µm. Są ruchliwe, z biegunowo umieszczoną rzęską i nieprzetrwalnikujące. Xanthomonas campestris pv. zeae daje ujemny wynik w teście na obecność oksydazy i dodatni w teście na obecność katalazy. Produkuje on żółty pigment – xantomonadynę. Na podłożu YDC jej kolonie są lekko żółte, lepkie i zawiesiste. Półselektywnym podłożem do izolacji tej bakterii jest podłoże MXP. Optimum wzrostu dla tego gatunku bakterii przypada na 28°C (Limura i Hosono 1995, Schaad i wsp. 2001).
Identyfikacja przy pomocy testów API 20E pozwoliła na wstępną identyfikację 71 izolatów bakteryjnych pochodzących ze zmienionych chorobowo miejsc na kukurydzy. Dla badanych izolatów bakteryjnych uzyskano następujące 3 rodzaje profilów metabolicznych wyrażonych za pomocą siedmiocyfrowego kodu API 20E (Tab. 1) co sugeruje dużą zmienność metaboliczną tego gatunku bakterii (Hartung i Civerolo 1991).
Tab. 1. Przykład wyników wstępnej identyfikacji bakterii z rodzaju Xanthomonas przy pomocy testów API 20E.
|
Izolat |
Pochodzenie |
Rodzaj |
Kod API 20E |
|
M081 |
Smolice |
7202000 |
|
|
M089 |
Smolice |
Xanthomonas |
2224400 |
|
M094B |
Smolice |
Xanthomonas |
5202000 |
Dla bakterii X. campestris istnieją na rynku gotowe zestawy do identyfikacji przy pomocy metod serologicznych. W badaniach izolatów bakteryjnych z liści kukurydzy, wstępnie zidentyfikowanych technikami biochemicznymi do rodzaju Xanthomonas użyto następujących zestawów do diagnostyki serologicznej: Do testu typu immunoblot – “Immunoblot kit, Alkaline phosphatase label, 1 membrane, plus kontrola pozytywana i negatywna “ (Agdia). Do testów aglutynacyjnych użyto zestaw “Express TM Spot Check LF plus kontrola pozytywna i negatywna“ (Neogen Europe). W literaturze opisano również test ELISA do wykrywania X. campestris (Alvarez 1985).
W wyniku przeprowadzonych badań przy pomocy zastosowanych surowic, w testach ELISA i immunoblot oraz w testach aglutynacyjnych wykryto szereg izolatów dających pozytywny wynik na obecność Xanthomonas campestris (Tab. 2.).
Tab. 2. Identyfikacja bakterii przy użyciu testów serologicznych.
|
Rodzaj użytego testu serologicznego |
Kierunek badania |
Izolaty, dla których zanotowano wynik dodatni dla danego testu
|
|
Immunoblot |
Xanthomonas campestris |
M081, M089, M094, M095, M108, M162, M193, M227, M242, M278, |
|
Testy aglutynacyjne |
Xanthomonas campestris pv. campestris |
M089, M094, M095, M108, M150, M156, M162, M190, M193, M197, M227, M242, M275, M311, M349, M625 |
Uzyskane wyniki testów serologicznych nie były zgodne z wynikami uzyskanymi za pomocą testów API. Wątpliwe przypadki w identyfikacji można potwierdzić poprzez sekwencjonowanie fragmentu genu 16SrRNA przy użyciu starterów uniwersalnych dla bakterii (Fig. 1 i Tab. 3.).
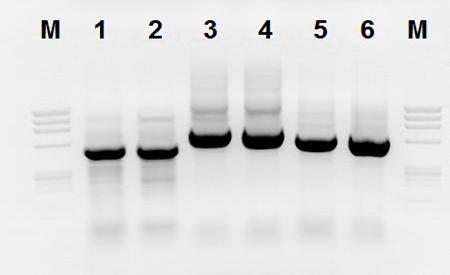
Fig.1. Produkty PCR trzech par starterów służących do namnażania 16S rDNA. Oznaczenia: Od lewej: linia 1 - marker DNA ØX174, linie 2 i 3 – produkt PCR pary starterów 16S01/pD2, linie 4 i 5 – produkt PCR pary starterów 16S02/pF2, linie 6 i 7 - produkt PCR pary starterów 16S04/pH2.
Tab. 3. Oligonukleotydy uniwersalne dla bakterii użyte w trakcie badań
|
Starter |
Region amplifikowany |
Sekwencja 5’à 3’` |
Ta (ºC) |
Długość produktu PCR (pz) |
Źródło |
Zastosowanie w badaniach |
|
16S01 |
16S rDNA |
AGTTTGATCCTGGCTCAG |
57 |
491 |
Gerischer i wsp. 2008 |
Namnażanie 16S rDNA |
|
pD2 |
16S rDNA |
GTA TTA CCG CGG CTG CTG |
57 |
491 |
Edwards i wsp. 1989 |
Namnażanie 16S rDNA |
|
16S02 |
16S rDNA |
CTCCTACGGGAGGCAGCAGT |
57 |
695 |
Gerischer i wsp. 2008 |
Namnażanie 16S rDNA |
|
pF2 |
16S rDNA |
ACG AGC TGA CGA CAG CCA TG |
57 |
695 |
Edwards i wsp. 1989 |
Namnażanie 16S rDNA |
|
16S04 |
16S rDNA |
AACTCAAAGGAATTGACGG |
63 |
596 |
Gerischer i wsp. 2008 |
Namnażanie 16S rDNA |
|
pH2 |
16S rDNA |
AAG GAG GTG ATC CAG CCG CA |
63 |
596 |
Edwards i wsp. 1989 |
Namnażanie 16S rDNA |
Literatura
Alvarez, A.M. 1985. Rapid identification of Xanthomonas campestris pv. campestris by Elisa. Plant Disease, 69: 1082-1086.
Claflin L. E. 2000. Diseases caused by Prokaryotes. In: White D. G. (ed.) Compendium of corn diseases, pp. 3-9. The American Phytopathology Society press.
Coutinho T. A., Wallis, F. M. 1991. Bacterial streak disease of maize (Zea mays L.) in South Africa. Journal of Phytopathology, 133: 112-120.
Gerischer U. 2008. Acinetobacter molecular biology. ISBN: 190 445 5204, liczba stron: 348.
Giester L. J., Rees J. M. 2004. Bacterial Diseases of Corn. Clay/Webster, St. Paul, MN, USA.
Hartung J. S., Civerolo E. L. 1991. Variation among strains of Xanthomonas campestris causing dims bacterial spot, Plant Diease, 7: 622-626.
Ignatov A., Sechler A., Schuenzel E. L., Agarkova I., Oliver B., Vidaver A. K., Schaad N. W. 2007. Genetic diversity in populations of Xanthomonas campestris pv. campestris in cruciferous weeds in central coast of California. Phytopathology, 97: 803-812.
Limura K., Hosono A. 1995. Biochemical characteristics of glucose-fermentative gram-negative bacteria isolated from buckwheat seeds. Curr. Advances Buckwheat Res. 127: 893–900.
Schaad N. W., Dianese J. C. 1981. Cruciferous weeds as a source of inoculum of Xanthomonas campestris in black rot of crucifers. Phytopathology, 71: 1215-1220.
Schaad N. W., Jones J. B., Chun W. 2001. Laboratory guide for identification of plant pathogenic bacteria. The American Phytopathological Society Press, Third Edition. St. Paul, Minnesota, USA.
