Pantoea agglomerans – Bakteryjna zaraza liści i więdnięcie naczyniowe kukurydzy i sorga (ang. leaf blight and vascular wilt of maize and sorghum).
Pantoea agglomerans – Bakteryjna zaraza liści i więdnięcie naczyniowe kukurydzy i sorga (ang. leaf blight and vascular wilt of maize and sorghum).
Występowanie i zakres gospodarzy
Gatunek ten jest polifagiczny i występuje powszechnie. Jego występowanie stwierdzono na uprawach kukurydzy w Meksyku i w Polsce, na obszarze Wielkopolski i Dolnego Śląska. P. agglomerans powoduje choroby kukurydzy, sorga, gryki i cebuli. Część jego szczepów jest niepatogeniczna dla roślin i wykorzystywana do biologicznej ochrony roślin przed chorobami. Gatunek ten może być również oportunistycznym patogenem człowieka.
Rozprzestrzenianie:
Pantoea agglomerans może wnikać do wnętrza roślin podobnie jak inne bakterie fitopatogeniczne przez naturalne otwory (aparaty szparkowe i hydatody), niesione kroplami wody oraz przez mikrouszkodzenia roślin powstające najczęściej w czasie silnych deszczy i wiatrów niosących cząsteczki brudu i drobinki piasku, jak również przez większe uszkodzenia roślin powstałe, np. w czasie gradobicia.
Szkodliwość:
Największe nasilenie tej choroby obserwować można w sezonach, w których panuje ciepła i deszczowa pogoda, czyli warunki optymalne do rozwoju bakterii fitopatogenicznych. Objawy chorobowe powodowane przez P. agglomerans są mało charakterystyczne i mają postać nieregularnych plam koloru słomkowego, rozciągających się na całą długość liścia, co istotnie zmniejsza możliwości asymilacyjne rośliny i przekłada się na niższy plon. Obecność tego patogena bakteryjnego na roślinie nie jest równoznaczna z rozwojem choroby. Bakterie należące do gatunków uznawanych za patogeniczne dla kukurydzy mogą na niej występować naturalnie i kolonizować powierzchnię liści i przestrzenie międzykomórkowe tej rośliny, nie powodując przy tym wystąpienia objawów.
 A
A  B
B
Fig. 1. Objawy chorobowe na kukurydzy cukrowej porażonej przez bakterię P. agglomerans w warunkach polowych.
Wykrywanie i identyfikacja
Do wykrycia obecności i identyfikacji P. agglomerans na uprawach kukurydzy w Polsce stosowano szereg metod. W metodzie biochemicznej w celu identyfikacji bakterii do rodzaju, dla 197 izolatów bakteryjnych dających dodatni wynik w teście patogeniczności na siewkach kukurydzy oraz w teście nadwrażliwości na tytoniu, wykonano oznaczenia biochemiczne opisane w Schaad i wsp. (2001). Oznaczenia te obejmowały: test na zdolność do fermentacji glukozy na pożywce Hugh-Leifson (HL), test na wytwarzanie pigmentu na pożywce z weglanem wapnia (YDC), wytwarzanie pigmentu fluoryzującego w świetle UV - test na pożywce King B. Wyniki tej identyfikacji dla rodzaju Pantoea przedstawione są w tabeli 1.
Tab. 1. Wyniki identyfikacji do rodzaju 197 izolatów bakteryjnych pozyskanych z kukurydzy i dających pozytywny wynik w testach patogeniczności.
|
Grupa feno-typowa |
Testy fenotypowe * |
Rodzaj |
Numery izolatów |
Liczba izo-latów |
|||
|
HL |
YDC |
KB |
D1M |
||||
|
I |
+ |
+ |
- |
- |
Pantoea |
M074, M129, M161, M241, M252A1, M252A2, M260, M297, M304, M331, M342, M389, M391, M394, M395, M400, M402, M404, M408, M412, M426, M431, M432, M433, M435, M436, M441, M443, M471, M593, M607, M631, M637, M642, M650, M653, M683, M692, M694, M700, M706, M711, M720, M734, M737, M741, M742, M782, M783, M786, M792, M803, M809, M813, M1185, M1345 |
56 |
HL-Fermentacja glukozy - Test na pożywce Hugh-Leifson
YDC-Wytwarzanie pigmentu na pożywce z węglanem wapnia - pożywka YDC
KB-Wytwarzanie pigmentu fluoryzującego w świetle UV - test na pożywce King B
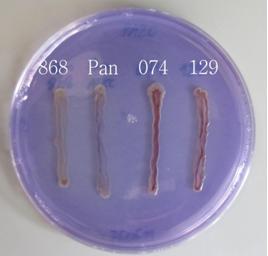
Przeprowadzone doświadczenie wykazało, że półselektywna pożywka PA20 dobrze różnicuje bakterie z rodzaju Pantoea w stosunku do bakterii należących do innych rodzajów (Rys. 2).
 A
A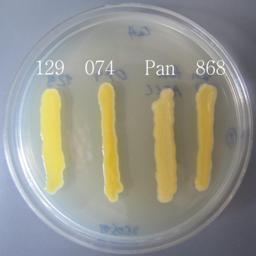 B
B
Fig. 2. Porównanie wzrostu izolatów bakteryjnych należących do gatunków P. ananatis (868 i Pan) oraz do gatunku P. agglomerans (074 i 129), na pożywkach PA20 (A) i TSA (B).
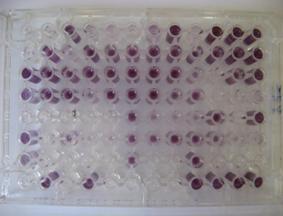
W celu identyfikacji do gatunku, izolaty oznaczone do rodzaju, badano przy użyciu komercyjnie dostępnych testów API. Wszystkie izolaty wymienione w tabeli 1 badano przy pomocy testów API 20E. Wyniki identyfikacji badanych izolatów, przy pomocy testów API, przedstawione są w tabeli 2 w postaci siedmiocyfrowego kodu określającego ich profil biochemiczny. W przeprowadzonym badaniu kontrolę pozytywną stanowiły kody API szczepów referencyjnych, zamieszczone w literaturze oraz baza danych programu API. Zastosowanie testów API pozwoliło na wstępną identyfikację 71 izolatów bakteryjnych. Dodatkowo izolaty te badano przy użyciu testu Biolog GEN III. Przykładowy wynik identyfikacji dla izolatu M260 przedstawiono w tabeli 2 i na rysunku 3.
Tab. 2. Identyfikacja wybranych izolatów bakteryjnych przy użyciu systemu BIOLOG.
|
Numer izolatu |
Wynik identyfikacji przy użyciu sytemu BIOLOG
|
Parametry systemu BIOLOG |
||
|
Prawdopodobieństwo (%) |
Podobieństwo |
Dystans |
||
|
M260 |
Pantoea agglomerans |
100 |
0,609 |
6,06 |
|
ATCC33243 |
Pantoea agglomerans |
100 |
0,598 |
6,04 |
 A
A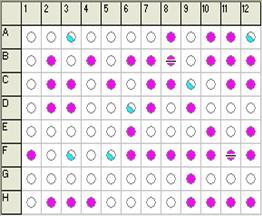 B
B
Fig. 3. Płytka identyfikacyjna systemu BIOLOG po inkubacji z izolatem M260 (P. agglomerans), gotowa do odczytu (A) oraz jej schematyczne przedstawienie w systemie BIOLOG (B).
Dodatkowo przeprowadzono również analizę filogenetyczną wybranych izolatów bakteryjnych należących do gatunków: P. ananatis, P. agglomerans i E. cloaceae subsp. dissolvens, w oparciu o sekwencję genu 16S rRNA (ok. 1500 pz). Do amplifikacji tego fragmentu genomu, używano równolegle 3 par starterów, których produkty PCR nakładają się na siebie. Otrzymane produkty PCR oczyszczono i poddano sekwencjonowaniu. Bazując na uzyskanych sekwencjach nukleotydowych genu 16S rRNA wybranych izolatów oraz na analogicznych do nich sekwencjach pobranych z GenBanku, przeprowadzono analizę filogenetyczną (Fig 4). Analizę przeprowadzono przy użyciu metody najbliższego sąsiada (ang. neighbor joining) w programie Mega 4.0. Jako wartość bootstrap przyjęto 1000 powtórzeń. Dane umożliwiające identyfikację izolatów są umieszczone na dendrogramach. Sekwencje genu 16S rRNA, uzyskane dla wybranych izolatów, po analizie bioinformatycznej zamieszczono w bazie danych GenBank (M260, GQ849221).
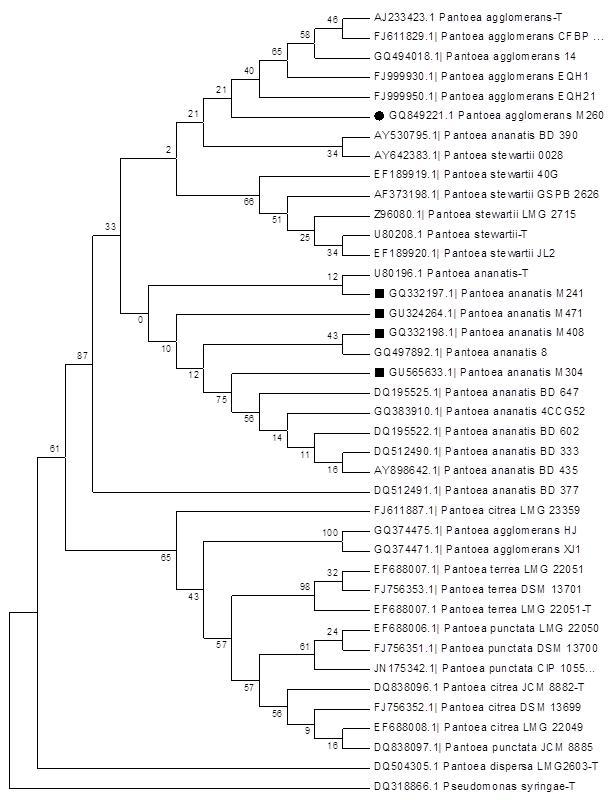
Fig. 4. Dendrogram przedstawiający analizę sekwencji genu 16S rRNA badanych izolatów zidentyfikowanych jako P. ananatis i P. agglomerans, skonstruowany metodą neighbor joining. W dendrogramie uwzględniono różne gatunki rodzaju Pantoea oraz 1 sekwencję Pseudomonas syringae (AF094749) użytą, jako grupa zewnętrzna. Wartość bootstrap wynosiła 1000 powtórzeń. Badane izolaty P. ananatis oznaczono na dendrogramie czarnymi kwadratami, natomiast badany izolat P. agglomerans oznaczono czarnym kołem. Sekwencje szczepów wzorcowych (ang. type strains) każdego analizowanego gatunku oznaczone są literą: t (-T), za nazwą gatunkową.
Przy pomocy opisanych wyżej metod udało się zidentyfikować 7 izolatów bakteryjnych P. agglomerans: M074, M129, M260, M331, M395, M404, M436.
Dodatkowo, dla potwierdzenia poprawności identyfikacji i odróżnienia od siebie blisko spokrewnionych gatunków bakterii: P. agglomerans i P. ananatis, opracowano startery specyficzne dla tych gatunków i mające zastosowanie w technikach: real-time PCR i HRM-PCR. Wyniki uzyskane przy pomocy tych technik przedstawione są na zdjęciach poniżej.
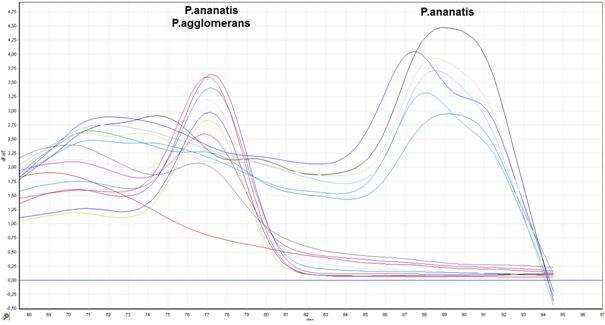
Fig. 5. Przebieg krzywych dysocjacyjnych (krzywych topnienia) produktów reakcji Real-Time PCR: Izolaty: P. ananatis: M241, M304, M408, M471, Pan 868, ATCC 33244; P. agglomerans: M074, M129, M260, M331, M395, ATCC 33243. Kontrola negatywna: kolor brązowy.
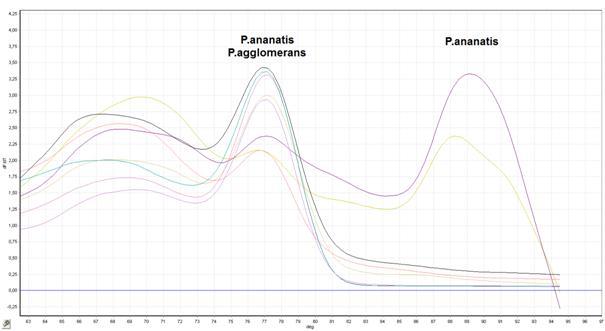
Fig. 6. Przebieg krzywych dysocjacyjnych (krzywych topnienia – Tm) produktów reakcji Real-Time PCR: Izolaty: P. ananatis: M304, ATCC 33244; P. agglomerans: M074, M395, M404, M436, ATCC 33243. Kontrola ujemna: kolor fioletowy
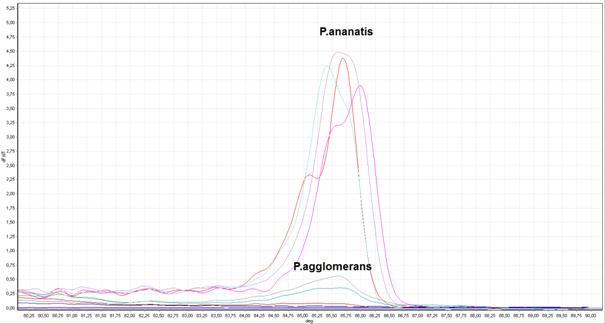
Fig. 7. Krzywe topnienia produktów reakcji HRM-PCR. Izolaty: P. ananatis: M241, M471, Pan868, ATCC 33244; P. agglomerans: M074, M129, M260, M331, M395. Kontrola ujemna – kolor brązowy.
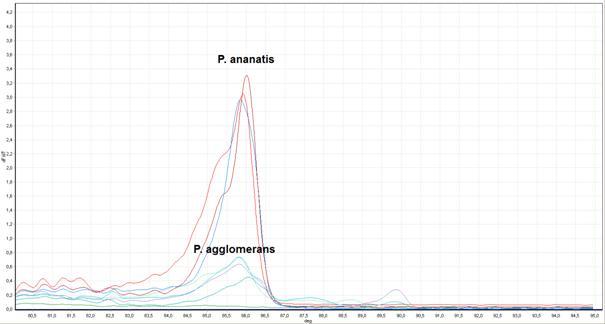
Fig. 8. Krzywe topnienia produktów reakcji HRM-PCR. Izolaty: P. ananatis: M304, ATCC 33244; P. agglomerans: M395, M404, M436. ATCC 33243. Kontrola ujemna – kolor zielony.
Ostatecznym potwierdzeniem identyfikacji badanych izolatów P. agglomerans było przeprowadzenie identyfikacji w oparciu o technikę MLSA. Wynik tej analizy przedstawiono na rysunku poniżej.
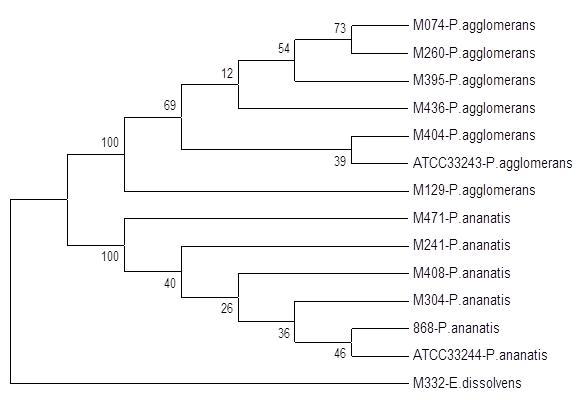
Fig. 9. Dendrogram przedstawiający analizę sekwencji genów: atpD, fusA rplB i rpoB, skonstruowany metodą najbliższego sąsiada (ang. neighbour joining) 13 izolatów z rodzaju Pantoea i jednego z rodzaju Enterobacter. Numery izolatów odpowiadają tym zebranym w tabeli 19. M332 – E. cloaceae subsp. dissolvens (grupa zewnętrzna); ATCC – izolaty referencyjne z kolekcji ATCC.
Przy pomocy analiz filogenetycznych użytych do badań, ustalono zaleznościfilogenetyczne między badanymi izolatami oraz wykazano, że przy pomocy techniki MLSA możliwe jest jednoznaczneodróżnenie od siebie izolatów bakteryjnych należących do gatunków P. ananatis i P. agglomerans. (Fig. 9).
Podsumowując, spośród badanych technik, do masowej detekcji P. ananatis przydatny będzie klasyczny PCR ze starterami PanITS1/EC5Gi i podłoże półselektywne PA20, które dobrze różnicuje gatunki P. ananatis i P. agglomerans od pozostałych bakterii kukurydzy. W przypadku niskiej koncentracji patogena doskonale sprawdzi się technika Bio-PCR z tą samą para starterów i podłożem półselektywnym PA20. Jako techniki rozstrzygające między gatunkami P. ananatis i P. agglomerans mogą być wykorzystane opracowane protokoły reakcji Real-Time PCR i HRM-PCR, natomiast w wyjątkowych przypadkach do ostatecznej identyfikacji badanego patogena zalecana jest technika MLSA.
